轻松搞定 R 语言包安装(最全攻略和常见错误解决方法总结)
简说基因-专业生信合作伙伴 2024-10-05 13:05:07 阅读 96
R语言包是扩展其功能的重要工具,涵盖数据处理、统计分析、图形展示等多个领域。如dplyr用于数据清洗和操作,ggplot2实现高级绘图等。
今天这篇文章我们来汇总一下R语言安装包的方式。
1.install.package() - 经典方式
1. 打开RStudio。
2. 在控制台中输入以下命令来安装包:install.packages("包名")。例如,如果要安装ggplot2包,可以输入install.packages('ggplot2')。
3. 按下Enter键运行命令。
4. R会自动连接到CRAN(Comprehensive R Archive Network)并下载安装所需的包及其依赖项。
5. 安装完成后,通过library(包名)命令加载已安装的包。例如,要加载ggplot2包,可以输入library(ggplot2)。
<code>install.package('ggplot2')
library(ggplot2)
这样,就可以在RStudio中成功安装和加载R包了。
2.Bioconductor - 生物信息学专属
很多的R包可能与与生物大数据分析及可视化相关,需要去网站Bioconductor,https://www.bioconductor.org/下载,也可以用BiocManager::install()方法安装。
1.首先,检查是否已安装BiocManager包,如果没有,可以使用以下代码安装:
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
2.然后,使用BiocManager::install()方法安装特定的Bioconductor包,例如安装clusterProfiler包:
BiocManager::install("clusterProfiler")
library(clusterProfiler)
3.devtools - 从 GitHub 安装
如果R包在GitHub上有开发版本,我们可以使用devtools包安装。
1.首先安装devtools包(如果尚未安装),然后运行 devtools::install_github("库名/包名")来安装。,GitHub网址:https://github.com/
# 安装并加载devtools
install.packages("devtools")
library(devtools)
devtools::install_github("hadley/ggplot2") # devtools::install_github("库名/包名")
2.也可以使用githubinstallR包,专门负责从GitHub安装R包,只需要提供R包名字,无需库名:
library(githubinstall)
install_github('dplyr')
4.手动安装 - 终极方案
手动安装方法一般用于前三种方法无法安装时,首先需要去官网下载自己需要的R包压缩文件。
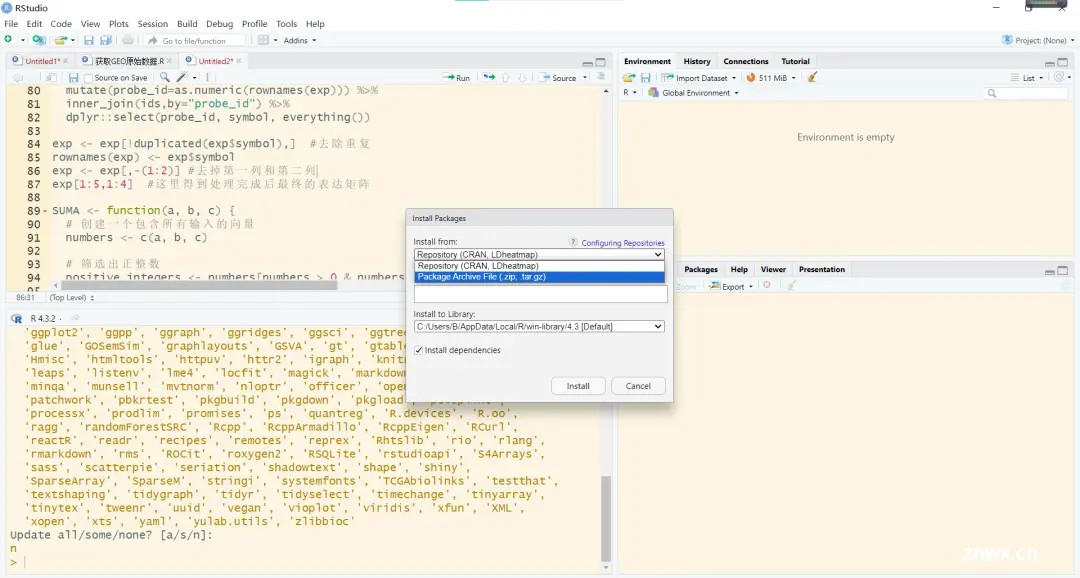
1.在Rstudio的主界面中找到Tools-->Install Packages-->Install from 找到R包压缩文件的位置并选中:

2.install.packages("path_to_package.zip", repos = NULL, type = "source")命令进行安装。:
<code>install.packages('R包所在的路径/R包文件名.tar.gz', repos= NLL, type='source')code>
5.R 包安装常见错误及解决方法
最后,我们再来总结一下安装R包过程中的常见错误。
• 包不可用错误:出现类似 "package '包名' is not available for this version of R" 的错误。
解决方法:尝试手动安装、使用BiocManager安装、使用devtools安装、降级R版本或寻找替代包。
• 依赖项错误:安装包时缺少依赖项导致失败。解决方法:安装缺少的依赖项。
####
#错误: package or namespace load failed for ‘clusterProfiler’:
# 'namespace:patchwork'没有出口‘patchworkGrob’这个对象
##需要安装patchwork包
install.package('patchwork')
library(patchwork)
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("clusterProfiler")
library(clusterProfiler)
或尝试使用install.packages("包名", dependencies = TRUE)来自动安装依赖项。
install.packages("clusterProfiler", dependencies = TRUE)
• 权限错误:没有足够的权限安装包到指定目录。
解决方法:以管理员身份运行R或将包安装到用户目录。
• 网络问题:由于网络连接问题无法下载包。
解决方法:检查网络连接,或手动下载包并本地安装。
• 包版本不匹配:包版本与R版本不兼容。
解决方法:查找适用于当前R版本的包或降级R版本。
• 包冲突:安装的包与已安装的包冲突。
解决方法:卸载冲突的包或使用install.packages("包名", lib = "指定路径")来解决依赖关系。
• 包路径问题:指定的包路径错误。
解决方法:确保路径正确,或使用install.packages("包名", lib = "指定路径")来指定安装路径。
• 其他问题:如编译错误、系统环境问题等。
解决方法:根据具体错误信息进行调查,可能需要安装额外的软件或库。
好了,关于 R 语言包的安装方式我们就总结到这里,大家有什么补充的欢迎在评论区留言。
一键分析10X单细胞数据(点击图片跳转)
一键分析Bulk转录组数据(点击图片跳转)

推荐阅读:
一文读懂scRNA-seq数据分析(建议收藏)
新年第一课:从零开始入门Galaxy生信云平台
经典教程:全转录数据分析实战
生物信息学中的可重复性研究
如何自学生物信息学:从菜鸟到专家
生信人的自我修养:Linux 命令速查手册
清华大学生物信息学课件资料分享
网上最全的 R 语言图库(建议收藏)| 简说基因 Recommend
生物信息学必备的R语言相关参考书 | 简说基因 Recommand
从单细胞数据分析的最佳实践看R与Python两个阵营的博弈
涉嫌侵权,容我解(jiao)释(bian)一下
关于简说基因
生信平台
Galaxy中国(UseGalaxy.cn)致力于打造中国人的云上生物信息基础设施。大量在线工具免费使用。无需安装,用完即走。活跃的用户社区,随时交流使用心得。
联系方式
QQ交流群(免费):925694514
微信交流群(免费):加微信好友,注明“Galaxy交流群”
客服微信:usegalaxy
声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。