Python | 实现 K-means 聚类——多维数据聚类散点图绘制
浅躬行 2024-07-21 14:05:01 阅读 88
文章目录
吐槽正题本文背景文章目的K-means 聚类步骤:K-means分类Python代码上述代码结果可视化展示不入流的小期待
吐槽
客观吐槽:CSDN的富文本编辑器真是超级无敌难用。首先要吐槽一下CSDN的富文本编辑器,好难用,好难用,好难用,好难用好难用,好难用,好难用,好难用!!!!!!!!!!!!!!!!!!前边的开头文字编辑了三四次,每次都是不小心按了ctrl+z,就完全消失了。
正题
本文背景
因需要想对某多维数据利用 K-means 聚类,并绘制散点图。几番查询发现,网上现有中文 K-means 介绍大多基于二维变量,在绘制散点图时直接以变量作为横纵最标轴即可,甚少有关于多维变量散点图的绘制说明。我涉算法不深,绘制该多维数据散点图颇费了些时间,特此记录,同时分享与大家,望能对同是入门小白的同学们有所帮助。
文章目的
利用 Python 自带包解决 K-means 多维数据聚类散点图绘制问题。
K-means 聚类步骤:
数据导入数据标准化变量相关性检验K 值的确定
1)手肘图或轮廓系数法。手肘图有基于平均离差法的,有基于SSE的,也有说可以直接从主成分/因子分析的碎石图判断的,我觉得几种方法本质上没啥区别,都以最明显拐点处的 K 值为准。
2)轮廓系数图通常以轮廓系数最大时的 K 值为准。
3)可以两者结合确定最佳的 K。聚类算法,确定数据的类别标签、聚类中心绘制散点图
1)若数据本身是二维的,直接以二维变量构建直角坐标系,为每类数据分配不同的颜色,绘制散点图即可;
2)若数据是多维的,绘制散点图之前需先将数据降维,平面图降为二维直角坐标系,立体图降为三维坐标系,并将聚类中心一并降维;将降维后的数据、聚类中心按照类别分配不同颜色,绘制在一张图中。
K-means分类Python代码
K-means 多维数据聚类上述所有流程如下,需求匹配度一致的同学可自取直用。
(期待:代码小白,不知道是否我的代码有问题,如果一次性直接运行,耗运行速度很慢,相当耗费时间。如果有大佬路过稍作指点,将不胜感激。)
(Tips:耗时最直接的解决办法,一个步骤一个步骤的运行,后续所有的步骤都基于标准化后的数据 df_normalized_data 数据集,且互相之间没有特别的相互影响,可运行完一步,注销掉相关代码,再运行下一步。)
<code>import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
from pandas import DataFrame
from sklearn import metrics
from sklearn.decomposition import PCA
# 导入数据,默认第一行为索引,index_col设定第一列也为索引
df_raw = pd.read_excel('data_SD_2.xlsx', sheet_name=0, index_col=0)
# 数据0-1标准化
df_normalized_data = df_raw.apply(lambda x: (x - np.min(x)) / (np.max(x) - np.min(x)))
# print(df_normalized_data)
# 利用皮尔逊相关系数查看多重共线性 及 可视化
df_corr = df_normalized_data.corr()
fig, ax = plt.subplots(figsize=(15, 10))
sns.heatmap(data=df_corr, annot=True, fmt='.2f', annot_kws={ 'size': 7}, cmap='Greens') # 若不喜欢绿色,可以通过cmap更改颜色code>
cax = plt.gcf().axes[-1]
cax.tick_params(labelsize=7)
plt.title('皮尔逊相关系数矩阵', fontsize=7)
plt.xticks(fontsize=7)
plt.yticks(fontsize=7)
plt.show()
# ----------------- 判断可以聚为几类:手肘图、轮廓系数法--------------------
# 手肘图法1——基于平均离差
K = range(1, 18)
meanDispersions = []
for k in K:
kemans = KMeans(n_clusters=k, init='k-means++')code>
kemans.fit(df_normalized_data)
# 计算平均离差
m_Disp = sum(np.min(cdist(df_normalized_data, kemans.cluster_centers_, 'euclidean'), axis=1)) / df_normalized_data.shape[0]
meanDispersions.append(m_Disp)
plt.rcParams['font.family'] = ['sans-serif']
plt.rcParams['font.sans-serif'] = ['SimHei'] # 使折线图显示中文
plt.plot(K, meanDispersions, 'bx-')
plt.xlabel('k')
plt.ylabel('平均离差')
plt.title('')
plt.show()
# 手肘图法2——基于SSE
distortions = [] # 用来存放设置不同簇数时的SSE值
for i in range(1,15):
kmModel = KMeans(n_clusters=i)
kmModel.fit(df_normalized_data)
distortions.append(kmModel.inertia_) # 获取K-means算法的SSE
# 绘制曲线
plt.plot(range(1, 15), distortions, marker="o")code>
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
plt.xlabel("簇数量")
plt.ylabel("簇内误差平方和(SSE)")
plt.show()
# 轮廓系数法
K = range(2, 10)
# 构建空列表,用于存储个中簇数下的轮廓系数
S = []
for k in K:
kmeans = KMeans(n_clusters=k)
kmeans.fit(df_normalized_data)
labels = kmeans.labels_
# 调用字模块metrics中的silhouette_score函数,计算轮廓系数
S.append(metrics.silhouette_score(df_normalized_data, labels, metric='euclidean'))code>
# 中文和负号的正常显示
plt.rcParams['font.sans-serif'] = ['Microsoft YaHei']
plt.rcParams['axes.unicode_minus'] = False
# 设置绘图风格
plt.style.use('ggplot')
# 绘制K的个数与轮廓系数的关系
plt.plot(K, S, 'b*-')
plt.xlabel('簇的个数')
plt.ylabel('轮廓系数')
# 显示图形
plt.show()
# ----------------- 开始 K-means 聚类的一系列过程---------------------
# K-means聚类
kms = KMeans(n_clusters=5, init='k-means++')code>
data_fig = kms.fit(df_normalized_data) # 模型拟合
centers = kms.cluster_centers_ # 计算聚类中心
labs = kms.labels_ # 为数据打标签
df_labels = DataFrame(kms.labels_) # 将标签存放为DataFrame
df_labels.to_excel('datalabels.xlsx') # 输出数据标签,其实输出可有可无
# 将聚类结果为 0,1,2,3,4 的数据筛选出来 并打上标签
df_A_0 = df_normalized_data[kms.labels_ == 0]
df_A_1 = df_normalized_data[kms.labels_ == 1]
df_A_2 = df_normalized_data[kms.labels_ == 2]
df_A_3 = df_normalized_data[kms.labels_ == 3]
df_A_4 = df_normalized_data[kms.labels_ == 4]
m = np.shape(df_A_0)[1]
df_A_0.insert(df_A_0.shape[1], 'label', 0) # 打标签
df_A_1.insert(df_A_1.shape[1], 'label', 1)
df_A_2.insert(df_A_2.shape[1], 'label', 2)
df_A_3.insert(df_A_3.shape[1], 'label', 3)
df_A_4.insert(df_A_4.shape[1], 'label', 4)
df_labels_data = pd.concat([df_A_0, df_A_1, df_A_2, df_A_3, df_A_4]) # 数据融合
df_labels_data.to_excel('data_labeled.xlsx') # 输出带有标签的数据
# 输出最终聚类中心
df_centers = DataFrame(centers)
df_centers.to_excel('data_final_center.xlsx')
# --------------------到这里 K-means 聚类的流程算是结束了------------------------
# ------------------------下面介绍如何绘制聚类散点图-----------------------------
# 对二分类的散点图绘制,网上教程很多,此篇文章主要介绍多分类的散点图绘制问题
# 首先,对原数据进行 PCA 降维处理,获得散点图的横纵坐标轴数据
pca = PCA(n_components=2) # 提取两个主成分,作为坐标轴
pca.fit(df_normalized_data)
data_pca = pca.transform(df_normalized_data)
data_pca = pd.DataFrame(data_pca, columns=['PC1', 'PC2'])
data_pca.insert(data_pca.shape[1], 'labels', labs)
# centers pca 对 K-means 的聚类中心降维,对应到散点图的二维坐标系中
pca = PCA(n_components=2)
pca.fit(centers)
data_pca_centers = pca.transform(centers)
data_pca_centers = pd.DataFrame(data_pca_centers, columns=['PC1', 'PC2'])
# Visualize it:
plt.figure(figsize=(8, 6))
plt.scatter(data_pca.values[:, 0], data_pca.values[:, 1], s=3, c=data_pca.values[:, 2], cmap='Accent')code>
plt.scatter(data_pca_centers.values[:, 0], data_pca_centers.values[:, 1], marker='o', s=55, c='#8E00FF')code>
plt.show()
上述代码结果可视化展示
皮尔逊相关系数图:

手肘图——基于平均离差:

手肘图——基于SSE:
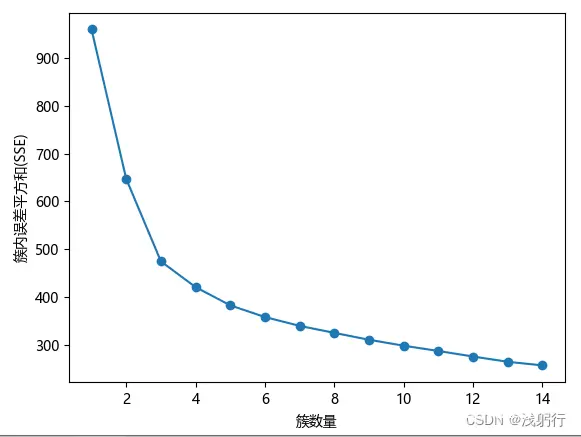
分析:从两个手肘图来看,拐点并不明显。硬要选一个的话,K = 3 是最佳选择。结合下面轮廓系数图也是K = 3。然而现实往往聚类的结果并不全部如所愿,因为根据现实情况理论上我的数据可以聚为5类(各种原因)。于是我是这样做的(科学性与否请大家自行判断,也特别期待大佬帮忙斧正):从轮廓系数图来看,K = 3 的确是最好的选择,然而K = 4和K = 5 的轮廓系数并没有明显区别,且后续在K > 5 后,轮廓系数又出现了非常明显的断崖式下降;结合手肘图中K = 3 和K = 5 的无论是SSE还是平均离差都有不小的降幅,最终选定K = 5。
轮廓系数图:(原图不便展示,下图来源于网络中趋势最接近的图片)

散点图绘制:(原图不便展示,下图来源于网络中趋势最接近的图片)

分析:多维数据的聚类散点图绘制,是需要将原始数据降维到二维或三维坐标系中的,降维方法本文选取的主成分方法,但其他方法同样可以使用。
希望所踩过的坑能发挥一些余热 . ~ _ ~ .
不入流的小期待
最后,非常不好意思推荐一下我的佛系小团子,希望看完文章有所收获的朋友们能动动小手帮我加个人气 . ~ _ ~ .
生活是生活,学习是学习,工作是工作,分享是分享。希望大家都学业顺利,生活快乐!

声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。