基于LIDC-IDRI肺结节肺癌数据集的放射组学机器学习分类良性和恶性肺癌(Python 全代码)全流程解析 (一)
雷博士 2024-06-13 12:35:02 阅读 87
基于LIDC-IDRI肺结节肺癌数据集的放射组学深度学习分类良性和恶性肺癌(Python 全代码)全流程解析 (一)
1 LIDC-IDRI数据集介绍1.1数据集预处理1.1.1 图像读取-(python库pylidc安装)1.1.2图像读取-(读取CT图像的patch和分割标注)1.1.3图像归一化-(读取CT图像的patch和分割标注) 1.2 图像标注信息预处理-(读取肺结节是良性还是恶性)1.2 肺结节图像切片处理处理-(切片并保存)1.3 总结第二部分传送门
第三部分传送门
1 LIDC-IDRI数据集介绍
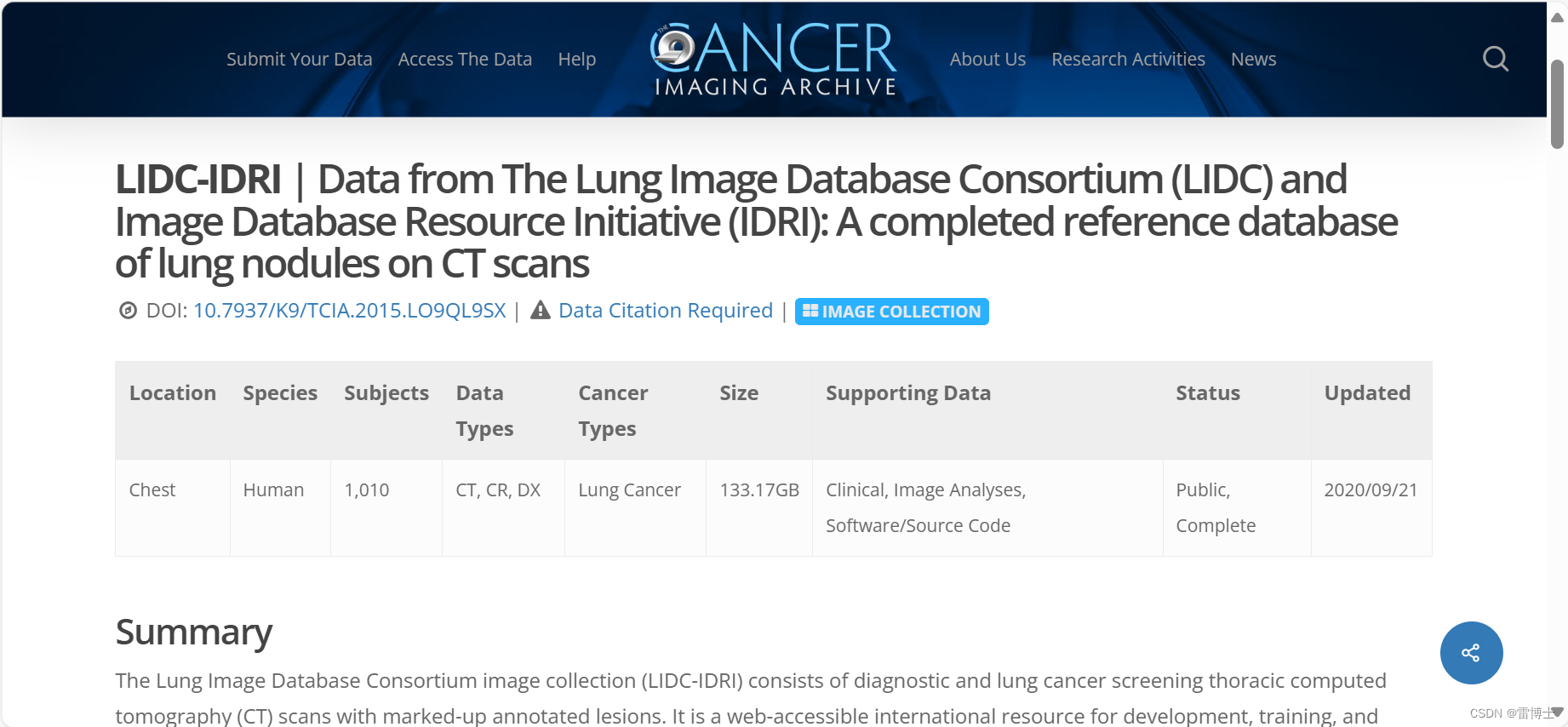
LIDC-IDRI数据集是用于医学影像分析的公开数据集,包含1010例低剂量CT扫描和人工标注的肺部结节信息。这些数据对于肺癌早期检测和算法开发至关重要。由美国国立卫生研究院(NIH)资助,该数据集为研究人员提供了宝贵资源,促进了医学影像处理和人工智能在肺部疾病诊断方面的进展,。
数据集下载地址如下:*https://www.cancerimagingarchive.net/collection/lidc-idri/*该数据集大小为133GB,数据集的下载需要下载器和稳定的网络,否则可能失败。
下载方面存在问题的私信我,我有完成数据集
1.1数据集预处理
数据集的预处理一共分为三个部分,图像的归一化,肺结节感兴趣区域分割,良恶性肺结节标注提取。
LIDC-IDRI数据结构如下:

1.1.1 图像读取-(python库pylidc安装)
在预处理图像过程中,我们不需要自己写程序去解析所有的文件。已经有人早就写好了免费的库文件去解析数据集。即为python的pylidc库。该库的安装如下:
打开anaconda prompt 输入

pip install pylidc
注意在安装完该库后,需要在系统用户文件夹下,新建pylidc.conf文件,为该库的运行指明文件存放的位置。
[dicom]
path = G:\dataset_zhang\LIDC-IDRI
将以上路径修改为数据集存放的路径。
测试该库是否安装成功代码如下:
import pylidc as plfrom pylidc.utils import consensusimport osdataset_path = r'G:\dataset_zhang\LIDC-IDRI\\'#修改路径哦dicom_name = 'LIDC-IDRI-0001'PathDicom = os.path.join(example) # 构建当前病例文件夹的完整路径# 查询当前病例的扫描数据,并将第一个扫描结果存储到scan变量中scan = pl.query(pl.Scan).filter(pl.Scan.patient_id == example).first()print(scan)
1.1.2图像读取-(读取CT图像的patch和分割标注)
我们以数据集中的’LIDC-IDRI-0001’病例数据为例,使用pylidc库中的按照病例文件夹名字的查询的方式,将该病例的所有的数据储存在scan中。而后提取所有专家对图像分割的标注坐标和肺结节的体素立方体。一个病例中含有多个肺结节默认提取第一个。使用的plt画图功能展示。代码如下
import numpy as npimport matplotlib.pyplot as pltimport matplotlib.animation as manimfrom skimage.measure import find_contoursimport cv2import pylidc as plfrom pylidc.utils import consensusimport osdataset_path = r'G:\dataset_zhang\LIDC-IDRI\\'dicom_name = 'LIDC-IDRI-0001'print(dicom_name) # 打印当前DICOM文件夹的名称PathDicom = os.path.join(dataset_path, dicom_name) # 构建当前DICOM文件夹的完整路径# 查询当前病例的扫描数据,并将第一个扫描结果存储到scan变量中scan = pl.query(pl.Scan).filter(pl.Scan.patient_id == dicom_name).first()vol = scan.to_volume()# 将该扫描数据转换成数组形式的体积(volume)# 聚类注释(nodule annotations)以获取一组注释nods = scan.cluster_annotations()try: anns = nods[0] # 尝试获取第一个注释(annotation) Malignancy = anns[0].Malignancy # 获取注释中的恶性程度(Malignancy)信息except IndexError: # 如果没有注释,或者无法获取第一个注释,则继续下一个DICOM文件夹 continue# 执行共识合并(consensus consolidation)和50%的一致性水平(agreement level)。# 我们在切片周围添加填充以提供上下文以进行查看。cmask, cbbox, masks = consensus(anns, clevel=0.5, pad=[(0,0), (7,7), (20,20)])# 提取相应的切片进行可视化image = vol[cbbox]k = int(0.5 * (cbbox[2].stop - cbbox[2].start))fig, ax = plt.subplots(1, 1, figsize=(5, 5))ax.imshow(vol[cbbox][:, :, k], cmap=plt.cm.gray, alpha=1)# 标记不同注释的边界colors = ['r', 'g', 'b', 'y']for j in range(len(masks)): for c in find_contours(masks[j][:, :, k].astype(float), 0.5): label = "Annotation %d" % (j+1) plt.plot(c[:, 1], c[:, 0], colors[j], label=label)# 绘制50%共识轮廓线for c in find_contours(cmask[:, :, k].astype(float), 0.5): plt.plot(c[:, 1], c[:, 0], '--k', label='50% Consensus')ax.axis('off') # 关闭坐标轴ax.legend() # 显示图例plt.tight_layout() # 调整布局以适应图像plt.show() # 显示图像
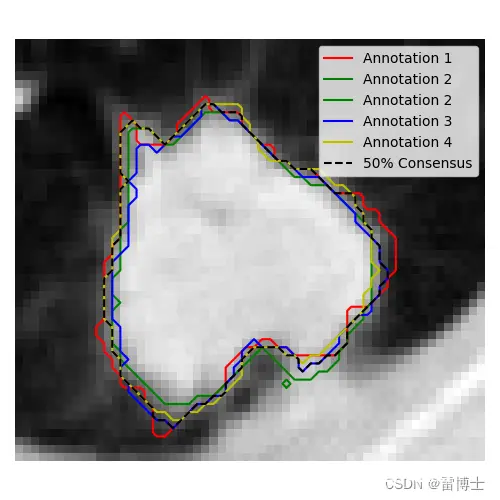
结果如下:
图片中,一共有4个专家对图像的分割的结果。和黑色的所有的专家对图像分割的平均的结果,一般情况下我们使用平均结果作为我们的实验参考。
1.1.3图像归一化-(读取CT图像的patch和分割标注)
图像归一化是一种将图像的像素值重新缩放到特定范围的处理方法,通常是将像素值映射到
0,1或者−1,1之间。这有助于提高模型的稳定性和收敛速度,使得不同图像具有相似的数据分布,有利于深度学习模型的训练和性能提升。代码如下
#归一化 def normalize_hu(image):#将输入图像的像素值(-4000 ~ 4000)归一化到0~1之间 MIN_BOUND = -1000.0 MAX_BOUND = 400.0 image = (image - MIN_BOUND) / (MAX_BOUND - MIN_BOUND) image[image > 1] = 1. image[image < 0] = 0. return image
1.2 图像标注信息预处理-(读取肺结节是良性还是恶性)
这段代码根据变量Malignancy的不同取值(如"Highly Unlikely"、"Moderately Unlikely"等),为label1赋予相应的标签(1到5)。通过这个过程,将文本标签映射为数字标签,方便后续机器学习模型的处理和训练。
if Malignancy == 'Highly Unlikely': label1 = 1 elif Malignancy == 'Moderately Unlikely': label1 = 2 elif Malignancy == 'Indeterminate': label1 = 3 elif Malignancy == 'Moderately Suspicious': label1 = 4 elif Malignancy == 'Highly Suspicious': label1 = 5 print(label1)
1.2 肺结节图像切片处理处理-(切片并保存)
首先根据image和cmask创建了ArrayDicom和ArrayDicom_mask矩阵。然后通过定位x_、y_、z_的中心点,对图像进行切片。接着将切片后的图像和对应的掩膜图像分别保存为大小为50x50的JPEG文件。同时,将图像的标签label1与文件名写入label.txt文件中。最后,将计数器ii增加,并输出计数器的值。
将处理后的图像进行切片和保存,并生成相应的标签信息。其中,ArrayDicom是图像数据矩阵,ArrayDicom_mask是对应的掩膜矩阵。通过对中心点定位,将图像在x、y、z方向上的切片进行大小为50x50的重采样和保存。同时,将每个图像切片的标签信息与文件名写入label.txt文件中,方便后续的数据处理和训练。
结果如下:
在label
这个label文件记录了每个图像文件的标签信息。每一行包含两个部分,以空格分隔:
第一部分是图像文件名,如 0.jpg, 1.jpg 等。
第二部分是对应图像的标签,标签是一个整数,表示图像的类别或者属性。在这个文件中,标签的含义可能是:
5: 高度可疑 (Highly Suspicious)
3: 不确定 (Indeterminate)
2: 中度可疑 (Moderately Suspicious)


1.3 总结
这样我们的就得到了我们需要训练的图像数据,它包括良性肺结节和恶性肺结节的2D图片和每一个肺结节的标签label。从三个不同方向上观察和处理图像,这在医学图像处理中是常见的。在这段代码中,每个方向的切片都被调整为大小为 (50, 50) 的图像,然后进行了插值以保持图像质量,并最终保存为 JPEG 图像文件。

“如果您有任何医学图像处理和机器学习项目需要技术支持,请随时私信我哦。”
有了这些文件我们就能够进行我们的放射组学特征的提取啦
完整代码如下:
import numpy as npimport matplotlib.pyplot as pltimport matplotlib.animation as manimfrom skimage.measure import find_contoursimport cv2import pylidc as plfrom pylidc.utils import consensusimport osdataset_path = r'G:\dataset_zhang\LIDC-IDRI\\'dicom_name = 'LIDC-IDRI-0001'#归一化def normalize_hu(image):#将输入图像的像素值(-4000 ~ 4000)归一化到0~1之间 MIN_BOUND = -1000.0 MAX_BOUND = 400.0 image = (image - MIN_BOUND) / (MAX_BOUND - MIN_BOUND) image[image > 1] = 1. image[image < 0] = 0. return image ii = 0# Query for a scan, and convert it to an array volume.for dicom_name in os.listdir(dataset_path): print(dicom_name) PathDicom = os.path.join(dicom_name) scan = pl.query(pl.Scan).filter(pl.Scan.patient_id == dicom_name).first() vol = scan.to_volume()# Cluster the annotations for the scan, and grab one. nods = scan.cluster_annotations() try : anns = nods[0] Malignancy = anns[0] Malignancy = Malignancy.Malignancy except : pass# Perform a consensus consolidation and 50% agreement level.# We pad the slices to add context for viewing. cmask,cbbox,masks = consensus(anns, clevel=0.5, pad=[(0,0), (7,7), (20,20)]) image = vol[cbbox] image = normalize_hu(image) k = int(0.5*(cbbox[2].stop - cbbox[2].start)) if Malignancy == 'Highly Unlikely': label1 = 1 elif Malignancy == 'Moderately Unlikely': label1 = 2 elif Malignancy == 'Indeterminate': label1 = 3 elif Malignancy == 'Moderately Suspicious': label1 = 4 elif Malignancy == 'Highly Suspicious': label1 = 5 print(label1) # 矩阵增广和传参 ArrayDicom = image ArrayDicom_mask = cmask # 中心点定位 x_, y_, z_ = np.shape(image) x_ = int(x_ / 2) y_ = int(y_ / 2) z_ = int(z_ / 2) # 图像进行切片处 save_dir = r'F:\test\data\train' # 修改为标签存放的位置 # 检查并创建文件夹 train_dirs = [ 'z', 'x', 'y'] for directory in train_dirs: full_dir = os.path.join(save_dir, directory) if not os.path.exists(full_dir): os.makedirs(full_dir) # label信息保存 txtfile = open(os.path.join(save_dir, 'label.txt'), mode='a') txtfile.writelines('%s %d \n' % (str(ii)+'.jpg', label1)) txtfile.close() # z方向切片 z_silc_50 = ArrayDicom[:, :, z_] z_silc_50 = cv2.resize(z_silc_50, (50, 50), interpolation=cv2.INTER_LINEAR) * 255 z_silc_50 = z_silc_50.astype(np.uint8) # 数据类型转换 cv2.imwrite(os.path.join(save_dir, 'z', str(ii) + '.jpg'), z_silc_50) # x方向切片 x_silc_50 = ArrayDicom[x_, :, :] x_silc_50 = cv2.resize(x_silc_50, (50, 50), interpolation=cv2.INTER_LINEAR) * 255 x_silc_50 = x_silc_50.astype(np.uint8) # 数据类型转换 cv2.imwrite(os.path.join(save_dir, 'x', str(ii) + '.jpg'), x_silc_50) # y方向切片 y_silc_50 = ArrayDicom[:, y_, :] y_silc_50 = cv2.resize(y_silc_50, (50, 50), interpolation=cv2.INTER_LINEAR) * 255 y_silc_50 = y_silc_50.astype(np.uint8) # 数据类型转换 cv2.imwrite(os.path.join(save_dir, 'y', str(ii) + '.jpg'), y_silc_50) ii += 1 print(ii)
请保护原创,转载不注明出处,将追究负法律责任!!!
声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。