药品包装或质量控制
CSDN 2024-08-29 08:31:31 阅读 83
下述代码是一个图像处理和机器学习分类的脚本,用于识别和分类药片。它使用了HALCON软件的命令集,HALCON是一个用于机器视觉和图像分析的高级编程语言和开发环境。
dev_close_window ()
read_image (Image, ‘blister/blister_mixed_reference’)
dev_open_window_fit_image (Image, 0, 0, -1, -1, WindowHandle)
set_display_font (WindowHandle, 14, ‘mono’, ‘true’, ‘false’)
dev_update_off ()
dev_display (Image)
dev_set_draw (‘margin’)
*
First, we extract the content of the blister and passthis information on to the gmm classifier
disp_message (WindowHandle, ‘Train gmm classifier on pill types’, ‘window’, -1, -1, ‘black’, ‘true’)
extract_pill_types (Image, Chambers, ChambersUnion, Classes, PhiRef, RowRef, ColumnRef, PillTypeCount)
NumClasses := |PillTypeCount|
create_class_gmm (3, 3, [1,5], ‘spherical’, ‘normalization’, 10, 42, GMMHandle)
add_samples_image_class_gmm (Image, Classes, GMMHandle, 0)
train_class_gmm (GMMHandle, 100, 0.001, ‘training’, 0.0001, Centers, Iter)Then, the subsequent blisters are tested for their right combination
Count := 12
for FileIndex := 1 to Count by 1
*
Align image read
read_image (Image, ‘blister/blister_mixed_’ + FileIndex$‘02’)
threshold (Image, Region, 90, 255)
connection (Region, ConnectedRegions)
select_shape (ConnectedRegions, SelectedRegions, ‘area’, ‘and’, 5000, 9999999)
shape_trans (SelectedRegions, RegionTrans, ‘convex’)
orientation_region (RegionTrans, Phi)
if (abs(Phi) > rad(90))
Phi := rad(180) + Phi
endif
area_center (RegionTrans, Area1, Row, Column)
vector_angle_to_rigid (Row, Column, Phi, RowRef, ColumnRef, PhiRef, HomMat2D)
affine_trans_image (Image, ImageAffinTrans, HomMat2D, ‘constant’, ‘false’)
reduce_domain (ImageAffinTrans, ChambersUnion, ImageReduced)
decompose3 (ImageAffinTrans, ImageR, ImageG, ImageB)Classify pill type for each chamber
classify_image_class_gmm (ImageReduced, ClassRegions, GMMHandle, 0.005) count_obj (ClassRegions, Number)
gen_empty_obj (FinalClasses)
connection (Chambers, ChambersRemaining)
for Index := Number to 1 by -1
dev_clear_window ()
select_obj (ClassRegions, Region, Index)
intersection (ChambersRemaining, Region, Region)
select_shape (Region, PillsOfOneType, [‘area’,‘width’], ‘and’, [200,40], [3000,68])
difference (ChambersUnion, PillsOfOneType, RegionDifference)
connection (RegionDifference, ConnectedRegions)
select_shape (ConnectedRegions, SelectedRegions, ‘area’, ‘and’, 0, 7868)
shape_trans (SelectedRegions, SelectedRegions, ‘convex’)
union1 (SelectedRegions, SelectedRegions)
difference (ChambersRemaining, SelectedRegions, ChambersRemaining)
concat_obj (SelectedRegions, FinalClasses, FinalClasses)
endfor
*
*
Check for right combination
gen_empty_obj (MissingPills)
gen_empty_obj (WrongPills)
gen_empty_obj (WrongNumberOfPills)
difference (ChambersUnion, FinalClasses, LeftOvers)
area_center (LeftOvers, Area, Row1, Column1)
if (Area > 0)
connection (LeftOvers, LeftOvers)
count_obj (LeftOvers, Number)
for Index := 1 to Number by 1
select_obj (LeftOvers, ObjectSelected, Index)
intensity (ObjectSelected, ImageB, Mean, Deviation)
if (Deviation > 40)
concat_obj (WrongPills, ObjectSelected, WrongPills)
else
concat_obj (MissingPills, ObjectSelected, MissingPills)
endif
endfor
endifCompute histogram
CountFinalClass := []
for Index := 1 to NumClasses by 1
select_obj (FinalClasses, ObjectSelected, Index)
connection (ObjectSelected, ObjectSelected)
count_obj (ObjectSelected, Size)
CountFinalClass := [CountFinalClass,Size]
endforDisplay classification results and output allover statistic
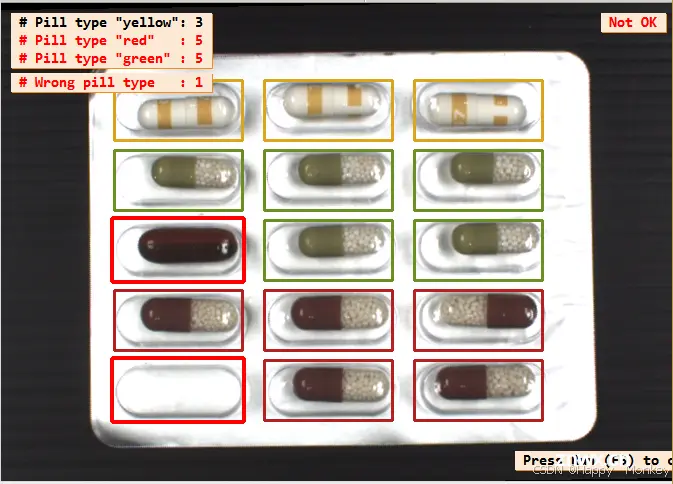
display_results (ImageAffinTrans, LeftOvers, FinalClasses, WrongPills, CountFinalClass, PillTypeCount, WindowHandle)
if (FileIndex < Count)
disp_continue_message (WindowHandle, ‘black’, ‘true’)
endif
stop ()
endfor Clear classifier handle
clear_class_gmm (GMMHandle)
blister_mixed_reference.png
blister_mixed_01.png
blister_mixed_02.png
blister_mixed_03.png
blister_mixed_04.png
blister_mixed_05.png
blister_mixed_06.png
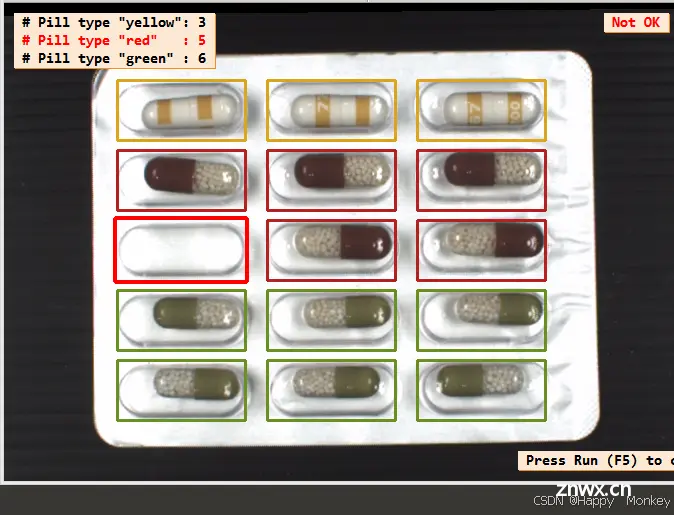
blister_mixed_07.png
blister_mixed_08.png

blister_mixed_09.png
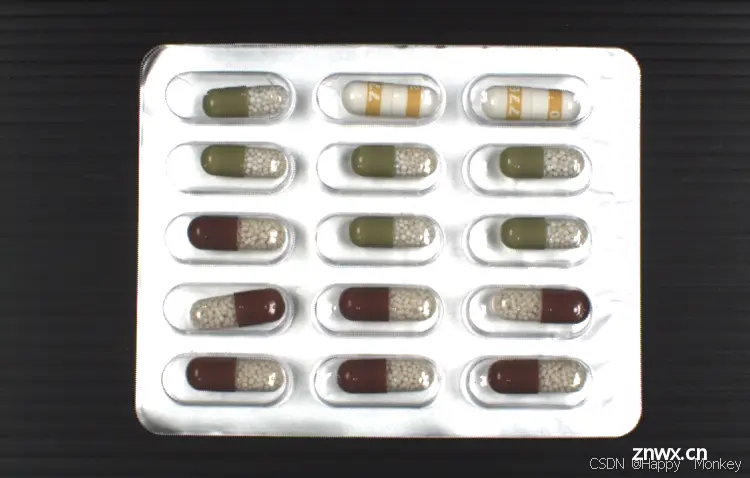
blister_mixed_010.png
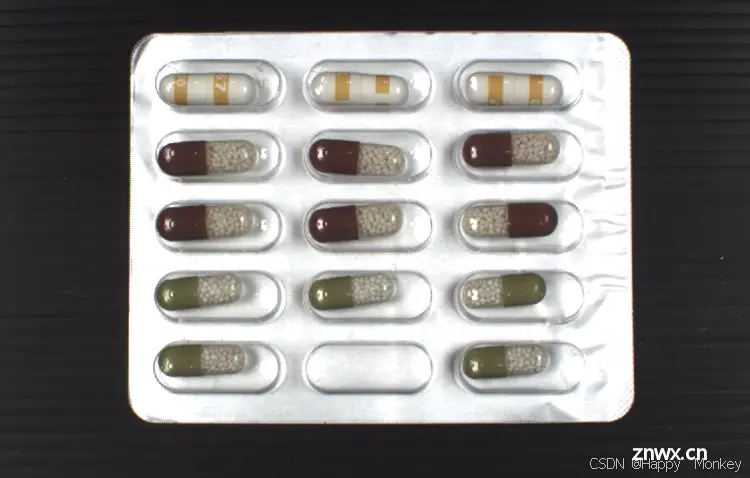
blister_mixed_011.png
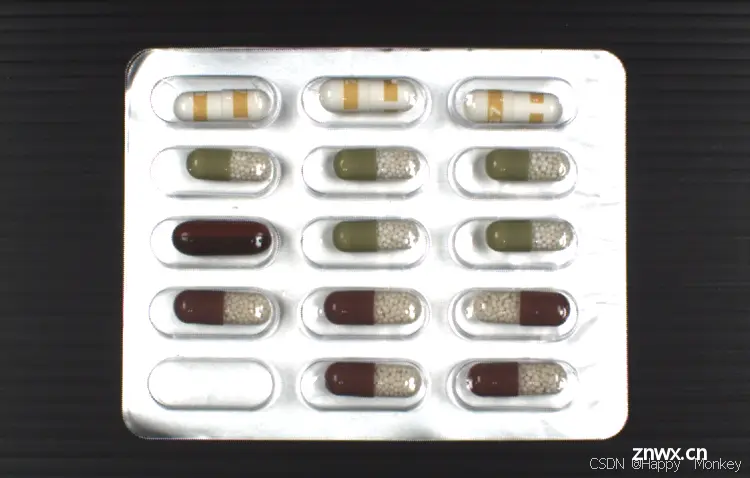
blister_mixed_012.png
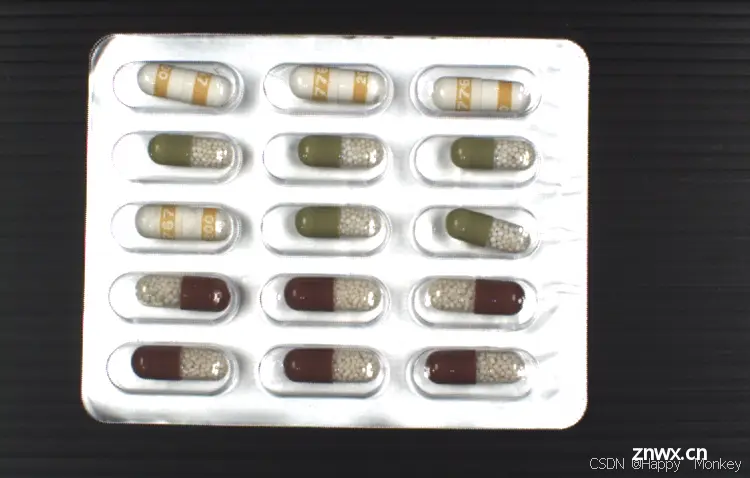
以下是程序运行结果:
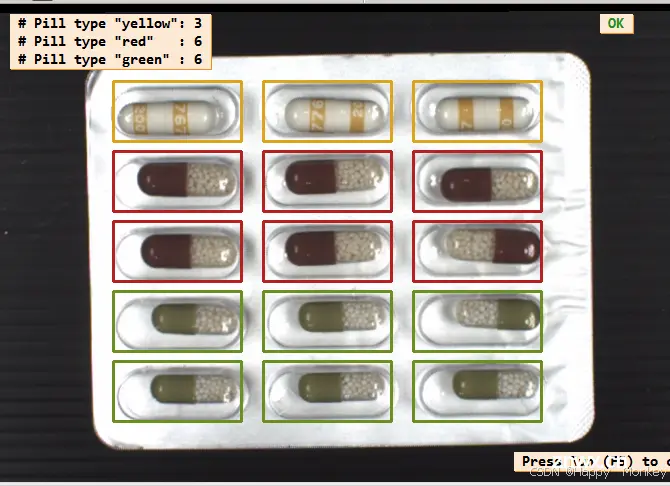
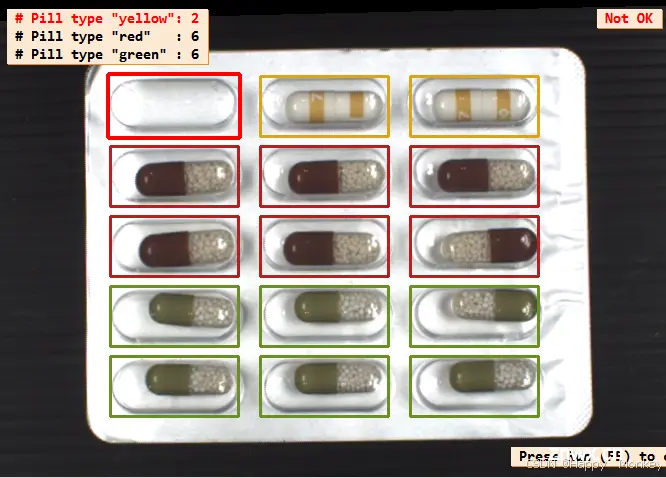

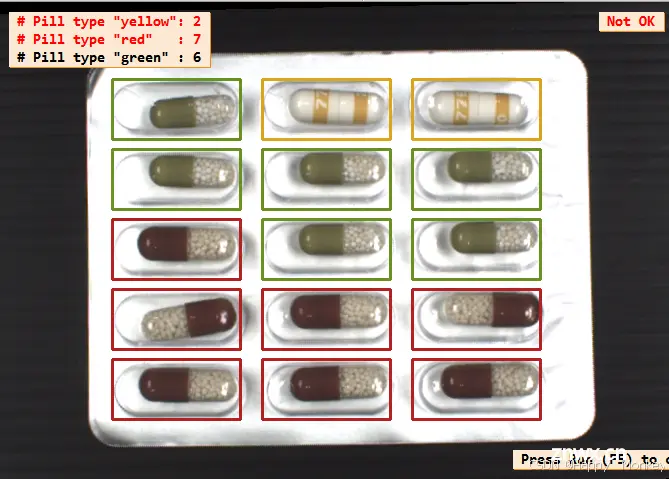
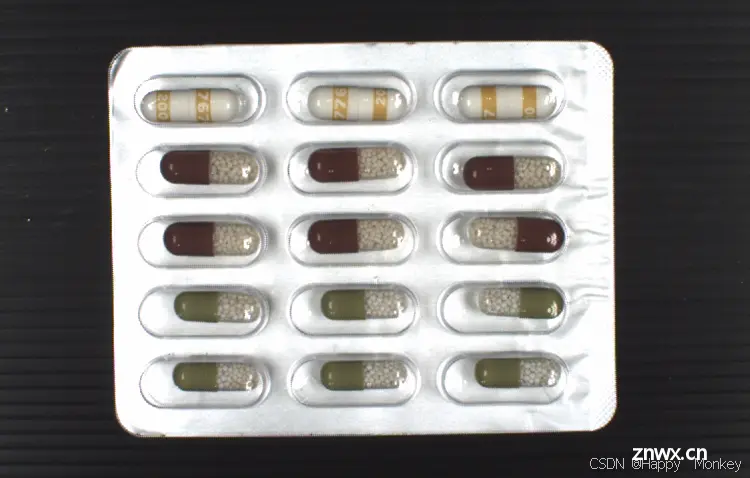
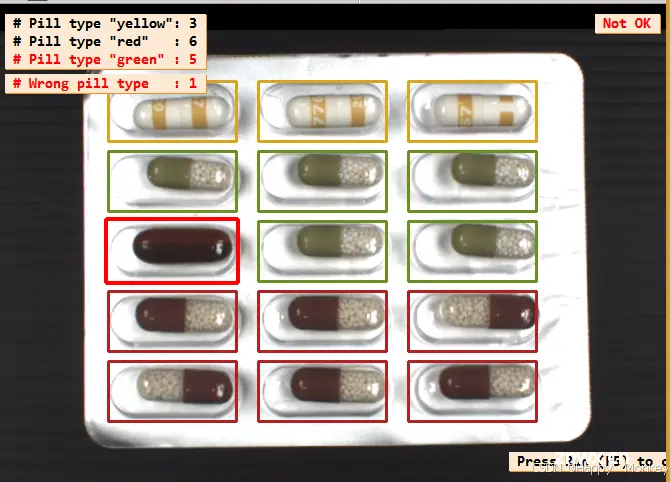
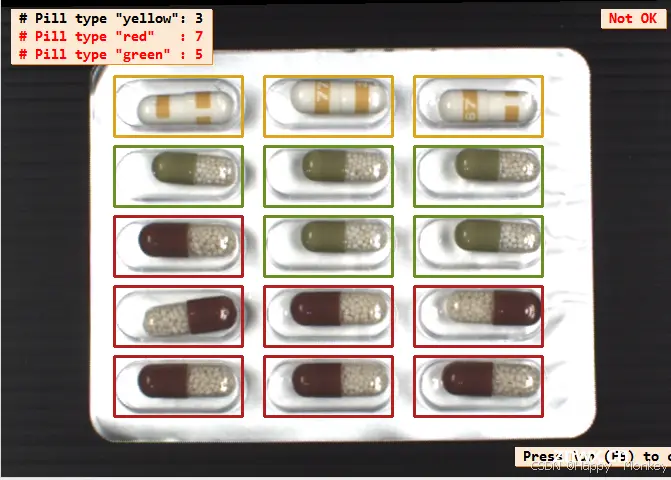


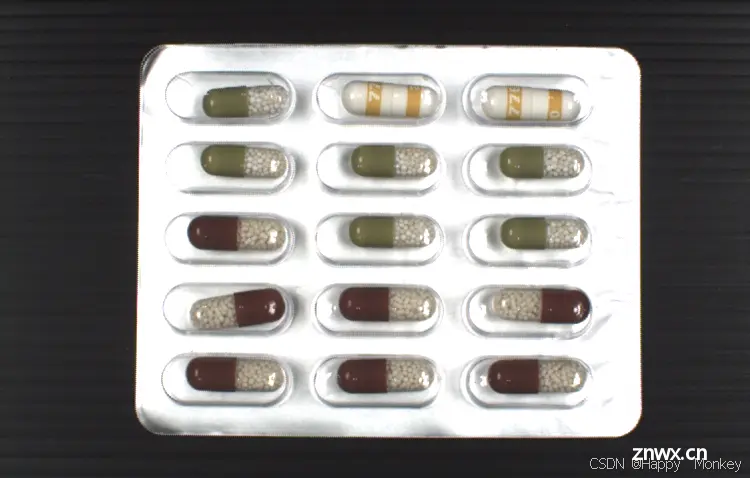
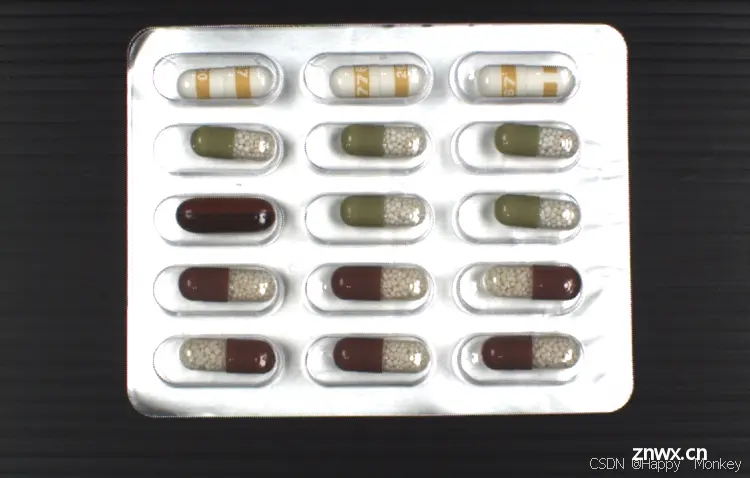

以下是代码的大致流程和功能解释:
初始化和显示设置:
dev_close_window():关闭当前窗口。
dev_open_window_fit_image():打开一个窗口以适应图像大小。
set_display_font():设置显示字体。
图像读取和预处理:
read_image():读取图像。
threshold():对图像进行阈值处理,提取感兴趣的区域。
connection():连接相邻的区域。
select_shape():根据面积筛选区域。
shape_trans():转换区域形状,例如转换为凸包。
orientation_region():计算区域的方向。
图像变换:
area_center():计算区域的中心点。
vector_angle_to_rigid():根据中心点和方向生成刚体变换矩阵。
affine_trans_image():对图像进行仿射变换。
颜色分解:
decompose3():将图像分解为红绿蓝三个颜色通道。
GMM分类器训练:
create_class_gmm():创建高斯混合模型(GMM)分类器。
add_samples_image_class_gmm():向GMM分类器添加图像样本。
train_class_gmm():训练GMM分类器。
药片类型分类:
classify_image_class_gmm():使用训练好的GMM分类器对图像进行分类。
后处理和统计:
count_obj():计算对象的数量。
gen_empty_obj():生成空的对象。
difference():计算差集。
union1():计算并集。
concat_obj():连接对象。
结果展示和统计输出:
display_results():展示分类结果和统计信息。
循环处理多个图像:
通过一个循环读取和处理多个图像文件。
清理资源:
clear_class_gmm():清理GMM分类器资源。
这段代码是一个完整的工作流程,从图像的读取、预处理、特征提取、分类器训练、分类、后处理到结果展示。它适用于自动化药片识别和分类的场景,例如在药品包装或质量控制中。
声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。