人工智能在病理切片虚拟染色及染色标准化领域的系统进展分析|文献速递·24-07-07
罗小罗同学 2024-10-20 16:01:01 阅读 67
小罗碎碎念
本期文献主题:人工智能在病理切片虚拟染色及染色标准化领域的系统进展分析
这一期文献的速递,是有史以来数量最大的一次,足足有十一篇,本来打算分两期写,但是为了知识的系统性,我决定咬咬牙,放在同一期推文里。
关于病理切片虚拟染色和染色标准化的研究,之前写过一期推文,但是我觉得还是不够系统,所以这里再补一期,不再局限于最新的文献,而是系统的回顾近三年的文献。从事这一方面研究的,大部分来自国内外顶尖的高校,例如加州大学洛杉矶分校、海德堡大学、清华大学、武汉大学……
另外,我还发现一个很有意思的现象,那就是感觉这个方向暂时停滞了(不排除我看的文献不够多),我觉得部分原因是方向可能偏了。要想把这个问题阐述清楚,我还需要把另外一个也遇冷的方向扯出来——泛癌分析。
这两个方向遇冷的原因,很有可能不是技术不行,而是数据收集不上来。虚拟染色/染色标准化+泛癌分析,可以极大的扩增数据量,并且也只有足够的数据量,足够的癌种的数据,才能学习到真实染色与虚拟染色之间的关系。各流程的规范性提升了,模型的泛化性也就提升了。

一、用于从未经染色的病理图像中进行分类和肿瘤定位的端到端深度学习框架

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Akram Bayat | 麻省理工学院媒体艺术与科学项目,媒体实验室 |
| 通讯作者 | Pratik Shah | 麻省理工学院媒体艺术与科学项目,媒体实验室 |
文献概述
这篇文章介绍了一个自动化的端到端深度学习框架,用于从未经染色的病理图像中进行分类和肿瘤定位。
研究由Akram Bayat、Connor Anderson和Pratik Shah等人完成,并发表在2021年SPIE医学成像会议的图像处理卷中。
背景与挑战:传统的组织病理学图像分析依赖于染色技术,但存在样本量少和染色过程不可逆等挑战。深度学习技术提供了一种计算虚拟染色的方法,以克服这些限制。研究目标:提出一个深度学习框架,能够直接从未经染色的前列腺活检图像(全切片图像,WSI)中自动检测和定位肿瘤。方法:
使用生成对抗网络(GAN)对未经染色的病理图像进行计算性H&E染色。利用卷积神经网络(CNN)在1024×1024像素的虚拟染色H&E图像块上检测和分类肿瘤区域。引入深度弱监督学习(WSL)模型,无需像素级注释即可定位用于肿瘤分类的主要组织学模式。 数据集与预处理:研究使用了来自38名患者的46对组织病理学切片图像,经过预处理后生成了95,605个图像块,用于训练、验证和测试。模型与实验:
实验评估了不同图像块大小对分类性能的影响,发现1024×1024像素大小的块最有效。研究了肿瘤和非肿瘤图像块在训练和验证数据中的分布比例,发现1.4的比例(非肿瘤:肿瘤)能够实现更高的分类性能。使用预训练的ResNet-18网络进行分类,并通过迁移学习优化模型性能。 结果:端到端系统在17K的测试集上评估,实现了86.37%的图像块分类准确率和85.05%的精确度,并达到了65.07±1.99的Dice指数。结论:该研究提出的深度学习框架能够自动化从组织染色到可解释的前列腺肿瘤分类的整个数字病理图像工作流程,对前列腺癌的准确分级和其他全切片图像分类任务具有潜在价值。
文章强调了深度学习在病理图像分析中的潜力,尤其是在减少对传统染色技术的依赖和提高诊断速度与准确性方面。
重点关注
Figure 7 展示了所提出的端到端框架如何在前列腺活检图像中定位肿瘤。
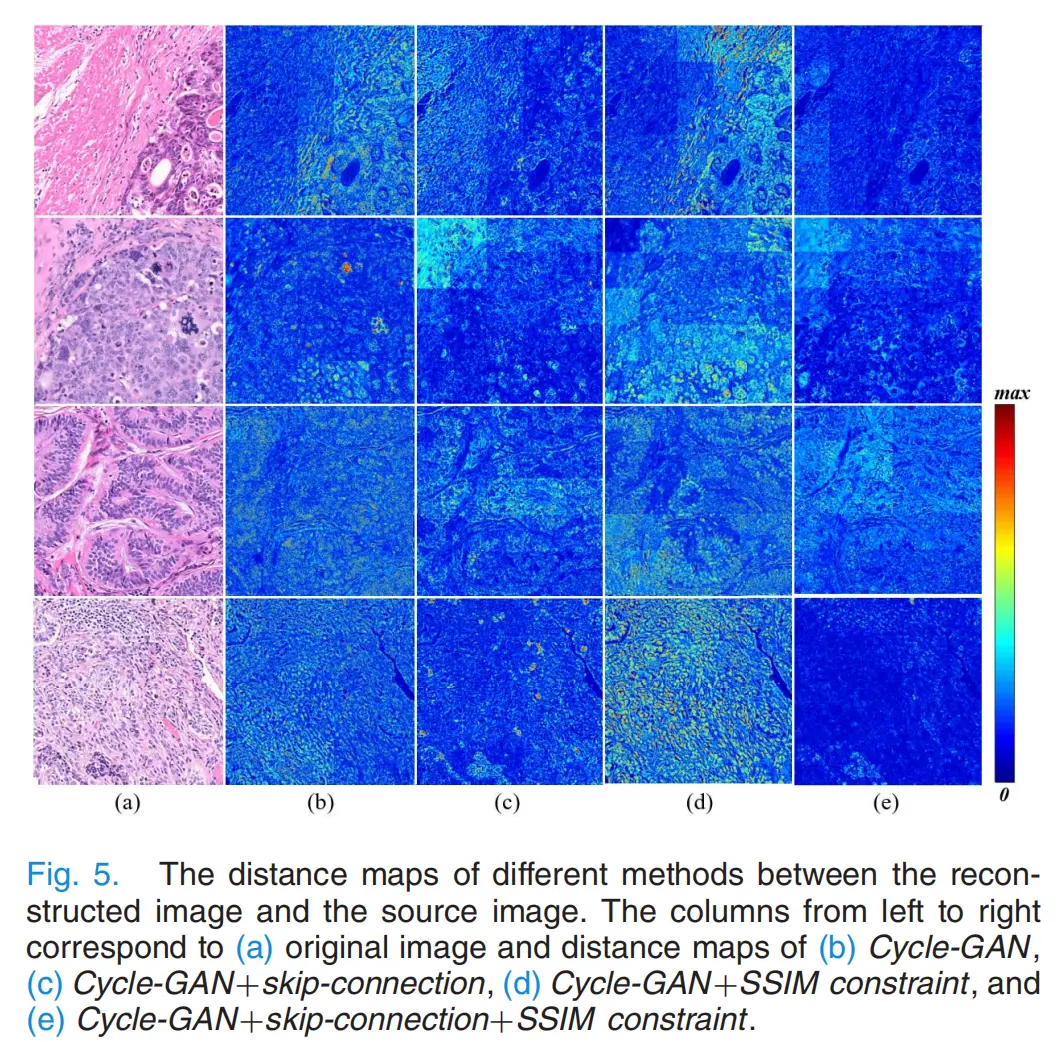
这个框架包括三个主要步骤,具体如下:
计算性H&E染色图像(a): 这是未经染色的原始病理图像,通过使用生成对抗网络(GAN-CS模型)进行计算性染色,转换成类似于传统H&E染色的图像。
梯度反向传播定位(b): 利用梯度反向传播(Gradient Backpropagation, GBP)方法,计算出图像中肿瘤区域的显著性图(saliency map)。GBP方法通过在网络的反向传播过程中添加来自高层神经元的激活信号,识别出对分类输出(即肿瘤分类)重要的输入图像像素。
叠加图像(c): 这是一个将定位结果叠加在原始图像上的可视化展示。在这张叠加图像中,强激活区域(即GBP定位出的肿瘤区域)用绿色显示,从而直观地表示出肿瘤的位置和范围。
Panels (d), (e), 和 (f) 按照与 (a), (b), © 相同的顺序展示了另一个样本图像的定位结果。这表明该框架能够应用于不同的图像样本,具有较好的泛化能力。
从临床角度来看,这种自动化的端到端框架可以为病理学家提供快速、准确的辅助诊断工具,帮助他们识别和评估肿瘤区域。此外,准确的肿瘤区域定位还可以为后续的治疗计划提供重要信息。
二、通过深度生成对抗网络(GAN)实现的虚拟卵巢癌组织染色方法

一作&通讯
| 角色 | 姓名 | 单位 | 单位(中文) |
|---|---|---|---|
| 第一作者 | 孟祥宇 | College of Computer Science and Technology, China University of Petroleum, Qingdao, Shandong, China | 中国石油大学计算机科学与技术学院,青岛,山东,中国 |
| 通讯作者1 | 李欣 | Department of Gynecology 2, Renmin Hospital of Wuhan University, Wuhan, Hubei, China | 武汉大学人民医院第二妇科,武汉,湖北,中国 |
| 通讯作者2 | 王勋 | China High Performance Computer Research Center, Institute of Computer Technology, Chinese Academy of Science, Beijing, China | 中国科学院计算技术研究所中国高性能计算机研究中心,北京,中国 |
文献概述
这篇文章是关于一种通过深度生成对抗网络(GAN)实现的计算虚拟组织染色方法(专门用于卵巢癌组织)。
这种方法旨在解决传统组织染色方法的复杂步骤和严格要求,这些因素严重影响了卵巢癌组织分析研究的进程。文章提出了一种基于GAN的弱监督学习方法,用以生成与卵巢组织的H&E(苏木精-伊红)染色切片相对应的未染色自荧光图像。通过医生评估,该方法生成的未染色荧光图像的准确度达到了93%。
研究者们还构建了一种虚拟染色过程的监督条件,使得后续虚拟染色阶段合成的图像质量更加完美。他们使用改进的UNet基础框架和提出的并行特征融合网络(PFFN),引入了更优秀的训练方法,以更好地适应模型达到最佳状态。与传统的图像翻译方法相比,改进后的图像翻译方法生成的图像质量更优。此外,研究者们还提出了一种特定于组织纹理的虚拟染色方法,并通过多种评估方法证实了其有效性。
文章还讨论了生成对抗网络(GAN)的相关研究进展,包括条件GAN、GAN的改进、图像到图像的翻译等。研究者们使用Wasserstein损失和特征匹配损失作为训练损失函数,并通过实验验证了这些损失函数在虚拟染色实验中的有效性。
最后,文章指出,这项研究提供了一种更高效的卵巢癌组织H&E染色解决方案,可以节省时间并快速协助医生进行诊断。研究还提出了一种有效的自荧光图像生成算法,可以在缺乏有效数据的情况下节省耗时的数据准备时间。未来的研究将扩展到更多类型的病理组织的虚拟染色,以实现更广泛的虚拟染色技术。
重点关注
Figure 7 展示了虚拟染色结果在卵巢癌病理切片上的显示。
虚拟染色图像与实际染色图像的比较:Figure 7 包含了由所提方法生成的虚拟染色图像与实际H&E染色图像的并排对比。这允许观察者直观地评估虚拟染色方法的效果。细节和特征的保留:虚拟染色图像应保留了与实际染色图像相似的细胞和组织细节,包括细胞核和细胞质的颜色区分,这对于病理学家进行诊断至关重要。颜色和对比度:虚拟染色图像的颜色和对比度应与实际H&E染色相似,H&E染色通常呈现为深紫色的细胞核和粉红色的细胞质。诊断价值:由于虚拟染色图像的目的是辅助病理诊断,Figure 7 中的图像需要展现出足够的病理特征,以便医生可以识别和分级癌症。评估准确性:根据文中提到的医生评估,虚拟染色的准确度达到了97%,这意味着在Figure 7中,大部分虚拟染色图像可能与实际染色图像在视觉上难以区分,且能够为病理诊断提供准确的信息。潜在的改进空间:尽管虚拟染色准确度很高,但可能仍有改进空间,比如在一些细节上可能需要进一步优化以完全匹配实际染色图像。
三、将非线性多模态(NLM)成像技术获取的图像转换成与常规的H&E染色相似的图像

一作&通讯
| 角色 | 姓名 | 单位名称(中文) | 单位英文名称 |
|---|---|---|---|
| 第一作者 | PRANITA PRADHAN | 弗里德里希-席勒大学物理化学研究所和光子学阿贝中心 | Institute of Physical Chemistry and Abbe Center of Photonics, Friedrich-Schiller-University |
| 通讯作者 | THOMAS BOCKLITZ | 耶拿大学光子学研究所和莱布尼茨健康技术研究所(成员单位) | Leibniz Institute of Photonic Technology, Member of Leibniz Health Technologies Jena |
文献概述
这篇文章是关于使用深度学习技术在生物医学光学领域的一项研究。
研究的核心是开发一种计算染色方法,将非线性多模态(NLM)成像技术获取的图像转换成与常规的Hematoxylin和Eosin (H&E)染色图像相似的图像。
H&E染色是组织病理学中的黄金标准方法,但它需要长时间的样本准备,限制了其在实时疾病诊断中的应用。为了解决这个问题,研究人员提出了一种无需染色的替代技术,即结合了相干反斯托克斯拉曼散射(CARS)、双光子激发荧光(TPEF)和二次谐波生成(SHG)三种非线性光学模式的NLM成像技术。
研究中使用了监督和非监督的深度学习模型,特别是条件生成对抗网络(CGAN)和循环条件生成对抗网络(cycle CGAN),来生成类似于H&E染色的伪彩色图像。这些模型的性能通过均方误差、结构相似性指数和颜色阴影相似性指数进行了定量分析。研究结果表明,这些深度学习模型在生成H&E图像方面表现出色,可以作为诊断应用中的有用工具,而无需执行基于实验室的染色程序。
此外,文章还讨论了训练数据集、图像预处理、目标函数和性能指标等因素对模型性能的影响,并提出了未来可能的研究方向,例如使用cycle CGAN模型进行多模态转换、图像增强和去噪等任务。
重点关注
图 1 展示了不同类型的图像,用于比较和评估通过不同方法生成的伪 H&E 染色图像。
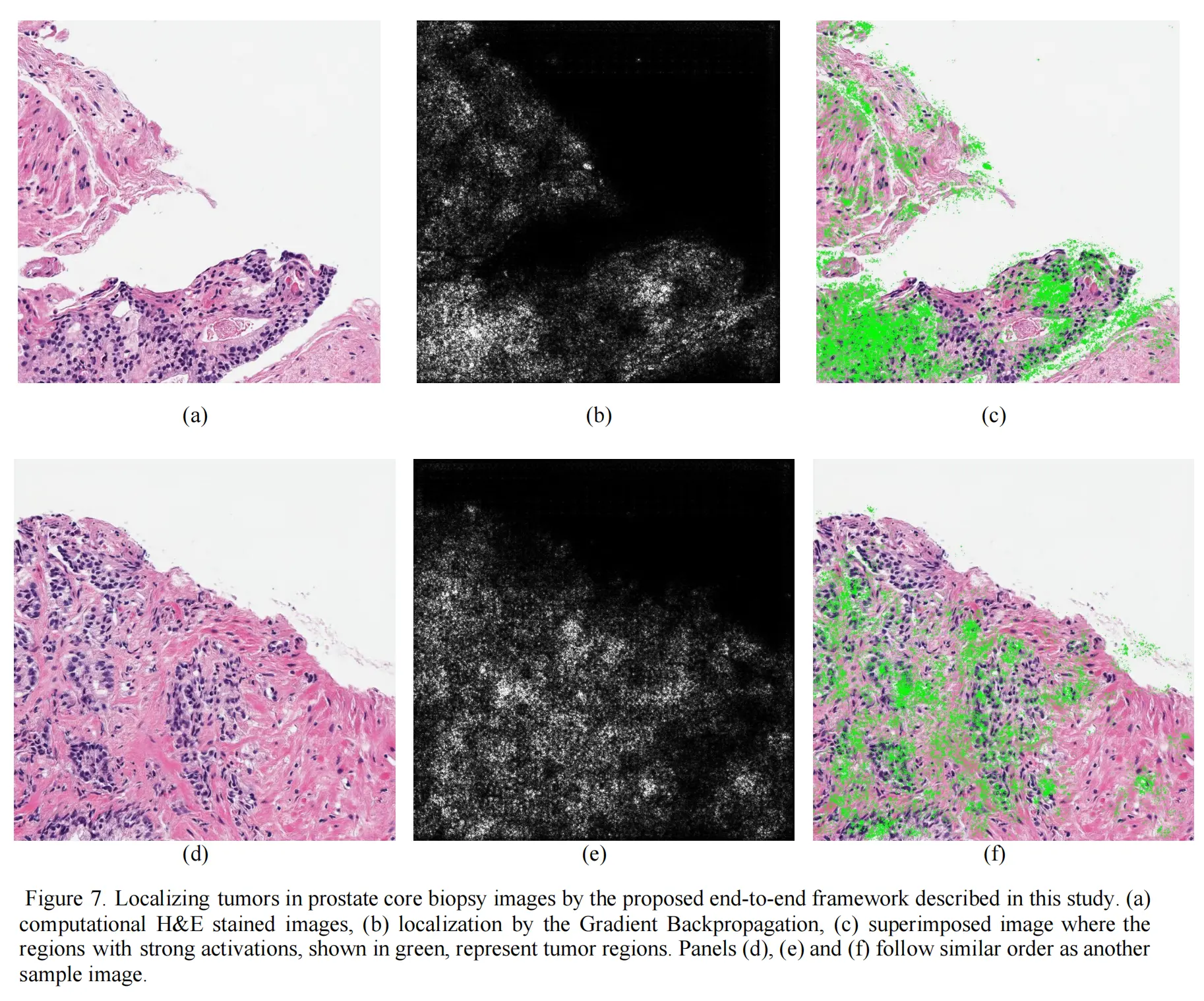
Fig. 1(A): 这是一个预处理过的非线性多模态(NLM)图像,其中CARS(相干反斯托克斯拉曼散射)、TPEF(双光子激发荧光)和SHG(二次谐波生成)分别作为红色、绿色和蓝色通道。这种图像是利用NLM成像技术获得的,可以提供生物组织的分子和结构信息。
Fig. 1(B): 显示了一个用于非监督伪 H&E 染色模型的组织病理学染色的 H&E 图像(或未配准的 H&E 图像)。在非监督模型中,不需要将 H&E 图像与 NLM 图像配准,因此这种图像被称为未配准的 H&E 图像。
Fig. 1©: 描述了一个配准的 H&E 图像,用于监督伪 H&E 染色模型。在这个图像中,配准效果通过用零填充显示出来,这表明了在配准过程中可能存在的空隙或不匹配的区域。
Fig. 1(D), (E), (F): 分别展示了使用监督方法、非监督方法以及参考文献 [12] 中使用的方法生成的计算染色 H&E 图像。这些图像模拟了传统的 H&E 染色效果,但它们是通过深度学习模型从 NLM 图像转换而来的。
所有图像都被缩小到原始大小的20%,以提高清晰度。所有图像中的尺度条代表了100微米,这为图像提供了尺寸参考。
从这个图表中,我们可以评估不同方法生成的伪 H&E 图像的质量,包括它们在色彩、结构和纹理方面与真实 H&E 染色图像的相似度。监督方法需要配准步骤,可能在图像质量上更接近真实的 H&E 图像,而非监督方法则不需要配准,可能在图像转换的自由度上更高,但可能在细节上有所损失。
四、使用深度学习技术将常规的H&E染色组织切片转换为特殊染色
一作&通讯
| 作者类型 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Kevin de Haan | 加州大学洛杉矶分校电气与计算机工程系 |
| 加州大学洛杉矶分校生物工程系 | ||
| 加州纳米系统研究所 | ||
| 通讯作者 | Yair Rivenson | 加州大学洛杉矶分校电气与计算机工程系 |
| 加州大学洛杉矶分校生物工程系 | ||
| 加州纳米系统研究所 | ||
| W. Dean Wallace | 南加州大学凯克医学院病理学与实验室医学系 | |
| Aydogan Ozcan | 加州大学洛杉矶分校电气与计算机工程系 | |
| 加州大学洛杉矶分校生物工程系 | ||
| 加州纳米系统研究所 | ||
| 加州大学洛杉矶分校医学院外科系 |
文献概述
这篇文章是关于使用深度学习技术将常规的H&E染色组织切片转换为特殊染色的研究。
研究的背景是病理学通常依赖于对组织切片进行视觉检查,而H&E染色是最常用的方法。尽管如此,特殊染色可以为不同的组织成分提供额外的对比度。研究团队展示了一种基于监督学习的计算方法,可以将H&E染色转换为特殊染色,使用肾脏穿刺活检组织切片进行演示。
研究结果表明,由三位肾脏病理学家进行评估,并通过第四位病理学家的裁决,从58位独特受试者获取的样本中生成的虚拟特殊染色图像,可以改善几种非肿瘤性肾脏疾病的诊断(P = 0.0095)。另一项研究发现,计算生成的特殊染色的质量在统计上与组织化学染色相当。这种染色转换框架可以在需要额外特殊染色时改善初步诊断,同时节省时间和成本。
研究还详细介绍了染色转换网络的设计和训练过程,包括使用深度神经网络来执行H&E染色组织和特殊染色之间的转换。通过使用单一的神经网络生成H&E图像以及特殊染色(PAS、MT和JMS),可以创建一个完全匹配的训练图像数据集。此外,为了确保网络能够处理H&E染色的不可避免的变异性,使用了八个独特的风格转换(染色标准化)网络来增强训练数据。
最后,研究通过盲法研究验证了所提出的染色转换技术,使用了来自58个不同H&E染色组织切片的全切片图像(WSI)数据,这些数据来自现有的非肿瘤性肾脏疾病数据库。研究结果显示,与仅使用H&E染色相比,使用附加的三个虚拟特殊染色可以显著改善多种非肿瘤性疾病的诊断结果(P = 0.0095)。通过使用染色转换技术,与使用组织化学染色的特殊染色相比,平均有15个初步诊断得到改善(25.8%),38.6个诊断一致(66.6%),4.3个诊断不一致(7.4%)。
文章还讨论了所提出方法的优势,包括减少化学处理、使用完美配对的训练图像对进行监督训练等。研究指出,这种方法可能有助于减少实际特殊染色的需求,节省时间和实验室费用,并可能在未来的诊断中发挥作用。研究使用了基于GAN的网络进行训练,并提供了详细的网络架构和训练过程描述。
重点关注
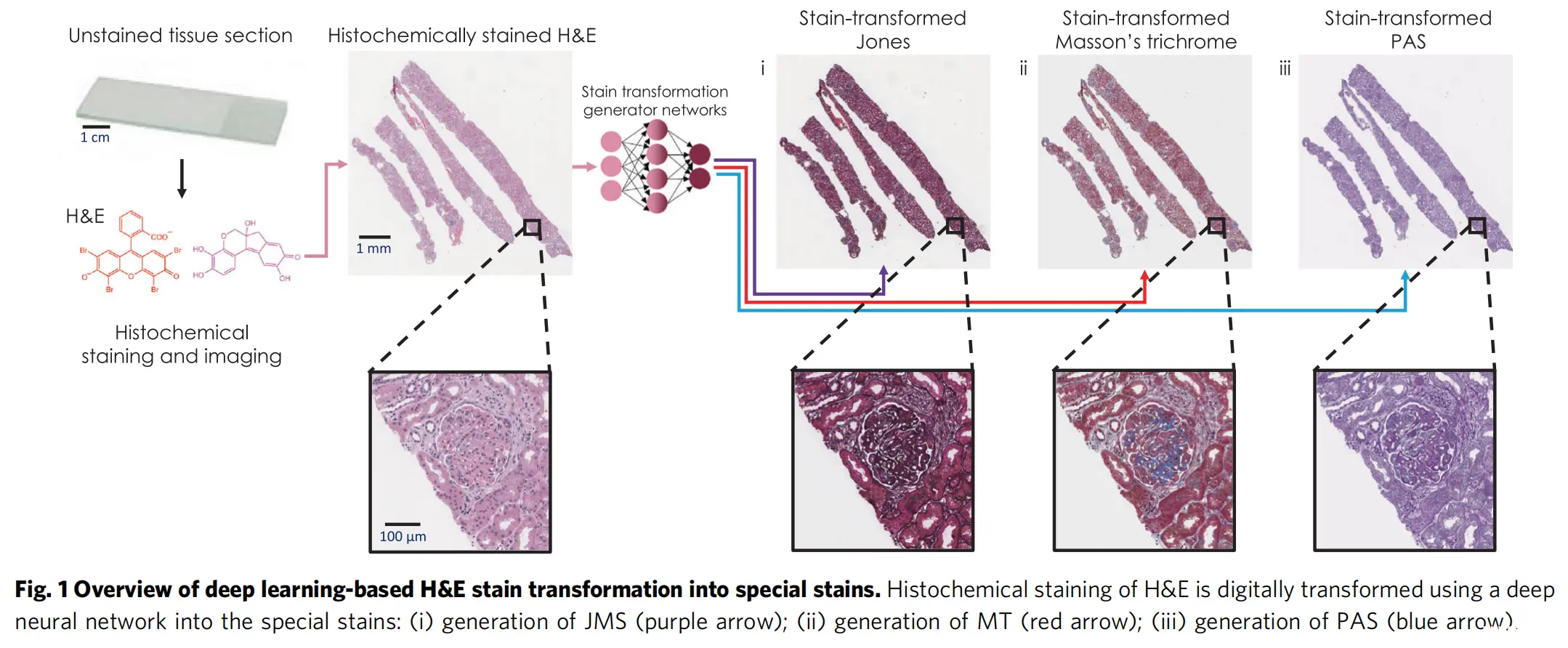
Fig. 1提供了深度学习基础的H&E染色转换为特殊染色的流程概览。

在这个过程中,常规的H&E染色组织图像通过深度神经网络被数字化转换为不同类型的特殊染色图像。具体来说,图中描述了三种特殊染色的生成过程,每种染色都用不同的颜色箭头进行了标记:
JMS(Jones methenamine silver)染色的生成:用紫色箭头表示。JMS染色是一种可以提供清晰对比,帮助病理学家观察肾小球结构和识别由于各种损伤引起的基底膜异常变化的染色方法。
MT(Masson’s trichrome)染色的生成:用红色箭头表示。MT染色用于观察结缔组织,能够提供组织结构的三色对比,帮助病理学家区分不同的组织成分。
PAS(periodic acid-Schiff)染色的生成:用蓝色箭头表示。PAS染色常用于更好地观察基底膜,通过染色可以揭示组织中多糖和蛋白质的结构。
此外,使用深度学习进行染色转换的优势在于,它能够减少实际的组织染色所需的时间和成本,同时避免了因多次染色而对组织样本造成的潜在损害。通过这种方式,可以快速生成多个虚拟染色图像,为病理学家提供更多的诊断信息。
五、医学图像分析中关于跨模态图像合成的研究
一作&通讯
| 角色 | 姓名 | 单位 | 单位(中文) |
|---|---|---|---|
| 第一作者 | Joel Honkamaa | Department of Computer Science, Aalto University, Finland | 芬兰奥卢大学计算机科学系 |
文献概述
这篇文章是关于在医学图像分析领域中,跨模态图像合成的研究。
跨模态图像合成是一个活跃的研究领域,具有多种临床应用价值。传统的方法要求训练数据对齐,但实际应用中获取对齐的数据对很困难。
本文提出了一种新的方法,允许使用成对但未对齐的训练数据进行训练,通过引入新的变形等变鼓励损失函数,该方法包括联合训练图像合成网络和单独的配准网络,并允许即使使用未对齐的数据也进行对抗性训练。
主要贡献和方法包括:
提出了一种新的架构,即使在训练数据未对齐的情况下也能稳健训练跨模态图像合成。通过模拟变形来鼓励网络学习到变形等变性,即在图像合成前后应用变形应得到相同的图像。开发了一种策略,允许在对抗性训练中根据输入图像进行条件化,这在医学领域尤为重要,因为它可以产生更可靠的预测。通过实验表明,该方法在非对齐数据上的表现优于早期方法,并且也超越了在训练前对图像进行配准的标准方法。
文章还详细介绍了所使用的损失函数,包括等变相似性损失、交换损失和对抗性损失,并通过一系列实验验证了所提出方法的有效性。
作者使用了不同的数据集进行评估,包括合成数据集、半合成的跨模态MRI合成数据集、虚拟组织学染色数据集和头部MRI到CT合成数据集。评估指标包括峰值信噪比(PSNR)、结构相似性指数(SSIM)、归一化互信息(NMI)和Fréchet inception distance(FID)。
重点关注
Fig. 9 展示了两个表现最佳的模型生成的虚拟染色组织学图像。这些图像展示了上皮细胞区域(粉红色)以及细胞核(蓝色)。
上皮细胞(粉红色):上皮细胞是覆盖在身体腔室和管道内表面的细胞,它们可以形成紧密的层以提供保护或分泌功能。细胞核(蓝色):细胞核是细胞的控制中心,包含遗传物质DNA。在组织学图像中,细胞核通常被染成蓝色,以便于识别和区分。在虚拟染色过程中,模型需要准确地识别和染色细胞核,这对于病理学分析至关重要。模型性能:文献中提到这两个模型是表现最佳的,这意味着它们在虚拟染色任务上达到了较高的准确度和真实感。这包括对细胞边界的清晰识别、正确的颜色渲染以及对细胞结构的精确模拟。应用意义:虚拟染色技术在数字病理学中非常有用,它可以减少对实际化学染色的依赖,加速病理学图像的分析过程,并可能提高诊断的准确性和效率。技术挑战:实现高质量的虚拟染色需要模型能够理解和模拟复杂的生物组织特性和染色过程。这包括处理图像中的不同纹理、结构和颜色分布,以及可能的噪声和变异。进一步研究:文档提到,尽管RegGAN模型在某些方面表现良好,但作者建议的配置(如EqSim + EqAdv 和 DefSim + Com + EqAdv)在更现实的数据集上表现更好。这表明在虚拟染色任务中,模型配置和损失函数的选择对最终结果有显著影响。
综上所述,Fig. 9 所展示的虚拟染色图像是对模型性能的一个直观展示,它们揭示了模型在理解和模拟复杂生物组织染色过程中的能力。
六、使用循环一致生成对抗网络(CycleGAN)对HE染色的组织学图像进行标准化

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Marlen Runz | 海德堡大学曼海姆医学院病理学研究所 |
| 通讯作者 | Cleo-Aron Weis | 海德堡大学曼海姆医学院病理学研究所及曼海姆智能医学系统研究所 |
文献概述
这篇文章是关于使用循环一致生成对抗网络(CycleGAN)对苏木精-伊红(HE)染色的组织学图像进行标准化的研究。
文章的背景是组织学图像在获取、组织处理、染色等方面存在显著差异,这些差异可能会阻碍图像分析,如染色强度评估或分类。为了减少这些差异,研究者们探索了图像标准化技术。
研究方法包括使用CycleGAN对临床数据进行颜色标准化,这些数据考虑了内部染色协议变化的变异性。CycleGAN由生成器网络GB和判别器网络DB组成,GB负责将图像从源域A映射到目标域B,而DB则训练以区分目标域B中的图像是真实的还是生成的。同样的过程也应用于另一对生成器-判别器对(GA和DA),用于反向映射GA: XB → XA。循环一致性确保生成的图像在反向映射时与其原始图像接近(GA(GB(XA)) ≈ XA,反之亦然)。
研究结果表明,通过将图像映射到目标域,来自该域的训练图像相似性提高了高达96%。同时,通过获得大于0.9的相似性指数,实现了生成器网络的高循环一致性。当将CycleGAN标准化应用于研究所的病理淋巴结数据时,仅在Camelyon16数据上训练的ResNet模型的kappa值增加了50%以上。
文章的结论是CycleGAN有效地标准化了HE染色图像,补偿了图像获取(如不同的扫描设备)和组织染色(如不同的染色协议)中的偏差,克服了来自不同机构的图像中的染色变异。
重点关注
Fig. 2 展示了构成 HEV 数据集的全切片图像(WSI)的示例缩略图,这些图像来自甲状腺组织的滤泡状癌细胞,并经过了 HE 染色。

每种染色方式都按照不同的染色协议进行了有意的调整:
A) 海德堡大学曼海姆医学院病理学研究所的标准染色协议(HE): 这是使用标准染色协议处理的组织切片,通常包含苏木精(Hematoxylin)和伊红(Eosin)两种染色剂,以显示细胞核和细胞质的不同特征。
B) 缩短染色时间(shortHE): 与标准染色相比,这一组切片的染色时间被有意缩短,可能会导致染色效果不均匀或某些组织结构的染色不足。
C) 延长染色时间(longHE): 这一组切片的染色时间被有意延长,可能会导致染色过深,影响对细胞细节的观察。
D) 仅用苏木精染色(onlyH): 这一组切片只使用了苏木精进行染色,通常用于强调细胞核和某些细胞结构的蓝色或紫色染色。
E) 仅用伊红染色(onlyE): 这一组切片只使用了伊红进行染色,通常显示为红色,主要用于突出细胞质和其他细胞结构。
这些不同的染色方式允许研究人员评估 CycleGAN 在不同染色条件下对 HE 染色图像进行标准化的能力。通过比较标准化前后的图像,可以评估 CycleGAN 在保持组织结构的同时调整染色强度和颜色的能力。这对于提高病理图像分析的准确性和一致性至关重要。
七、用于病理切片染色归一化处理的区域真实感知生成对抗网络

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Elif Baykal Kablan | 卡拉登尼兹技术大学,软件工程系,特拉布宗,土耳其 |
文献概述
这篇文章是关于一种新型的生成对抗网络(GANs)方法,称为区域真实感知生成对抗网络(RRAGAN),用于染色归一化处理。
染色归一化是计算机辅助诊断(CAD)系统中标准化颜色显示的一种技术。该技术对于维持组织和细胞结构的完整性以及颜色信息至关重要。
文章首先介绍了染色归一化的重要性和现有方法的局限性。然后,作者提出了RRAGAN模型,该模型扩展了CycleGAN,利用了PatchGAN判别器的输出,称为区域真实感知掩模(Regional Realness-Aware Mask),以指导生成器关注输入图像中的哪些区域是不正确的。这样,生成器在接下来的迭代中更加关注这些区域,从而产生更逼真的图像。
RRAGAN的性能在常用的MITOS-ATYPIA组织病理学数据集上进行了评估,并与五种最先进的方法进行了比较。结果显示,RRAGAN在峰值信噪比(PSNR)、结构相似性指数(SSIM)和均方根误差(RMSE)等指标上取得了显著的优势。此外,作者还评估了RRAGAN对MICCAI’16 GlaS数据集上分割性能的影响,发现它通过减少染色颜色变化,将分割性能提高了4.3%。
文章还讨论了RRAGAN模型的架构和训练方法,以及在两个数据集上的实验设置和结果。最后,作者得出结论,RRAGAN作为一种预处理步骤,可以显著帮助CAD系统在颜色变化下展示稳定的性能,并且可以作为处理数字病理学中颜色变化的一个潜在工具。
重点关注
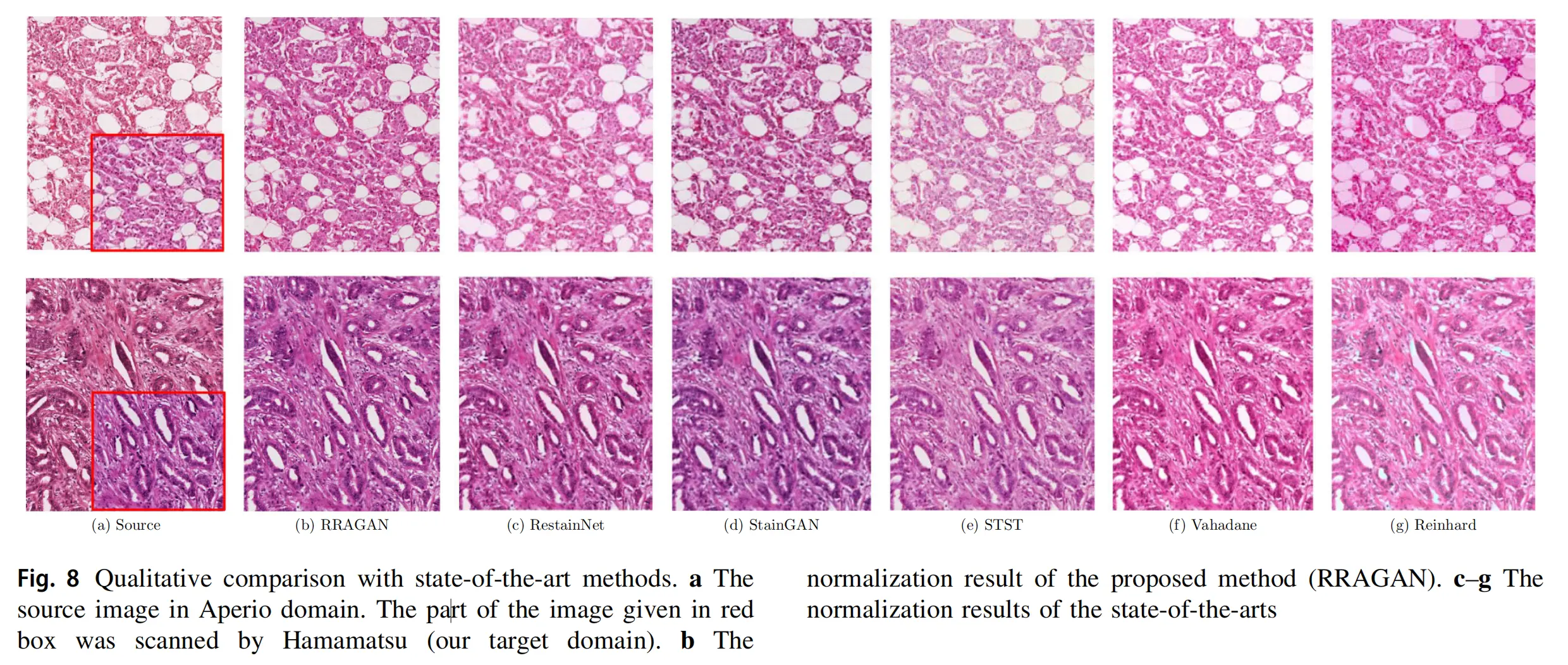
Fig. 8 展示了使用不同染色归一化方法后的定性比较结果。

a. 源图像(Aperio域): 这是原始的染色图像,由Aperio扫描仪扫描得到。图中用红框标出的部分是Hamamatsu扫描仪扫描的图像,作为目标域,即我们希望归一化后达到的效果。
b. 提出的方法(RRAGAN)的归一化结果: 这是使用本文提出的RRAGAN方法处理后的图像。根据文章的描述,RRAGAN应该在保持结构细节的同时,有效地将Aperio域的图像颜色转换为目标域(Hamamatsu)的颜色样式。
c–g. 现有最先进方法的归一化结果: 这些图像分别展示了使用其他五种最先进方法(Reinhard、Vahadane、StainGAN、STST、RestainNet)进行染色归一化的结果。每种方法都试图将源图像的颜色分布转换为目标图像的颜色分布。
在分析这些图像时,我们应该注意以下几点:
颜色转换的准确性: 归一化后的图像是否成功地模仿了目标域的颜色样式,同时保持了原始图像的颜色信息。
结构信息的保持: 在颜色转换的过程中,图像中的细胞和组织结构是否得到了保留,没有出现失真或模糊。
伪影和噪声: 归一化过程中是否引入了伪影或噪声,这可能会影响后续的图像分析和诊断。
整体视觉效果: 归一化后的图像在视觉上是否与目标图像相似,颜色是否均匀一致。
文章中提到,RRAGAN在多个评价指标上优于其他方法,包括峰值信噪比(PSNR)、结构相似性指数(SSIM)和均方根误差(RMSE)。这意味着RRAGAN在图像质量、结构相似性和误差最小化方面表现更好。定性分析(Fig. 8)应该能够支持这些定量结果,展示RRAGAN在视觉上更接近目标域图像,同时保持了良好的结构细节和较低的噪声水平。
八、使用残差循环生成对抗网络改善病理组织切片的图像分析

一作&通讯
| 角色 | 姓名 | 单位名称(中文) |
|---|---|---|
| 第一作者 | Thomas de Bel | 荷兰Radboud大学医学中心病理科 |
| 通讯作者 | Jeroen van der Laak | 荷兰Radboud大学医学中心医学图像科学与可视化中心 |
文献概述
这篇文章是关于使用残差循环生成对抗网络(Residual CycleGAN)来改善病理组织切片的图像分析的研究报告。
背景:在病理学中,不同医疗中心之间的染色变化是常见的,这可能对机器学习算法的可靠性产生显著影响。方法:提出了一种使用残差循环生成对抗网络(CycleGAN)的方法,通过学习染色变化的残差来减少性能的变异性。这种方法在常规CycleGAN的基础上加入了残差学习。实验:作者首先在一个单一来源中心的组织切片上训练了一个分割模型,同时大量应用增强技术以提高对未见数据的鲁棒性。然后,他们评估并比较了在其他中心的数据上应用CycleGAN染色转换的分割性能,包括有无转换的情况。结果:在结肠组织分割任务中,作者的方法在整体Dice系数上比非标准化目标数据提高了9%,比传统染色转换提高了4%。在肾脏分割任务中,残差CycleGAN在没有转换的情况下提高了10%的性能,与非残差CycleGAN相比提高了约2%。结论:残差CycleGAN通过学习源域和目标域之间的残差映射,能够专注于领域适应,同时保持结构完整性。这使得它在处理染色变化时特别有效,并且在医学图像分析中具有潜在的应用价值。贡献:作者在文中还比较了他们的方法与其他方法(包括传统染色转换和颜色增强技术),并涵盖了来自五个中心的数据,涵盖了两个不同领域的具有挑战性的分割任务。未来工作:尽管作者展示了残差CycleGAN在染色转换中的有效性,但他们也指出了需要进一步研究的地方,例如在临床设置中如何决定是否需要染色转换,以及如何进一步减少转换过程中可能出现的伪影。
重点关注
Fig. 3 展示了来自五个不同中心的结肠组织样本的 H&E(苏木精-伊红)染色切片。

这些样本的展示目的在于揭示不同医疗中心之间在染色方面的高度多样性。
多样性展示:图中的样本显示了即使使用常规的 H&E 染色协议,不同中心的染色结果也可能有显著差异。这种差异可能来源于组织固定和处理、染色程序、切片厚度等多种因素。
染色质量:H&E 染色的质量直接影响了病理学家在显微镜下对组织切片的诊断。不同中心的染色质量差异可能会对病理诊断的一致性和准确性造成影响。
数字化影响:随着全切片扫描仪的普及和计算机辅助诊断系统的流行,组织切片的数字化引入了新的变异源。例如,扫描时的设备设置、分辨率和存储格式等都可能影响最终的数字图像。
对算法的影响:染色的多样性对深度学习算法在医学图像分析中的性能有显著影响。深度学习系统对染色变化非常敏感,这要求算法不仅要能够处理结构信息,还要能够适应风格的变化。
研究意义:这项研究通过展示不同中心染色的多样性,强调了在开发和部署病理图像分析算法时,需要考虑染色标准化或转换的重要性。这对于提高算法的泛化能力和减少对特定染色条件的依赖至关重要。
后续应用:文章中提出的残差循环GAN方法,正是为了解决这种染色多样性带来的挑战。通过学习染色之间的残差映射,该方法能够改善染色变化对分割算法性能的影响,提高算法在不同染色条件下的鲁棒性。
Fig. 3 的展示是本文研究的出发点之一,它直观地说明了为什么需要对染色过程进行标准化或转换,以及这些技术如何帮助提高病理图像分析的准确性和可靠性。
九、研究不同神经网络架构对于虚拟染色的影响,并系统评估模型可行性

一作&通讯
| 角色 | 姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Umair Khan | 芬兰图尔库大学,生物医学研究所 |
| 通讯作者 | Pekka Ruusuvuori | 芬兰图尔库大学,生物医学研究所 芬兰坦佩雷大学,医学与健康技术学院 FICAN West 癌症中心,癌症研究单位,图尔库大学医院 |
文献概述
这篇文章的主要研究了不同的神经网络架构对于虚拟染色的影响,并系统评估了这种方法在组织学上的可行性。
文章的主要亮点包括:
展示了基于深度学习的虚拟染色技术,使用常规的明场显微镜图像。证明了增加网络容量可以产生更好的虚拟染色效果。该方法具有广泛的适用性,并且有潜力减少对化学染色的需求。
研究使用了生成对抗网络(GAN)模型pix2pix的变体,对来自多个器官的组织样本进行了定量和视觉评估,以比较虚拟染色与H&E染色的真实样本的复制精度。
研究发现,通过使用密集卷积单元替代简单的卷积,可以提高结构相似性得分、峰值信噪比和细胞核复制精度。此外,研究还展示了网络架构优化可以提高虚拟H&E染色的图像转换精度,突出了虚拟染色在简化组织病理学分析中的潜力。
文章还讨论了虚拟染色技术在临床病理学中的应用前景,特别是在癌症诊断中。研究表明,通过增加神经网络的复杂性,可以显著提高虚拟染色的质量,减少伪影,并且能够更准确地再现细胞和亚细胞结构的细节。
最后,文章指出,虽然虚拟染色技术在某些方面已经显示出比化学染色更高的准确性,但在解释如亚核形态等细节时仍需谨慎。研究还强调了网络容量的增加对于提高虚拟染色质量的重要性,并指出了未来研究的方向,包括对不同组织类型进行特定评分以提高结果的解释性,以及探索在不同病理任务中使用标准明场成像设置和图像分辨率的潜力。
重点关注
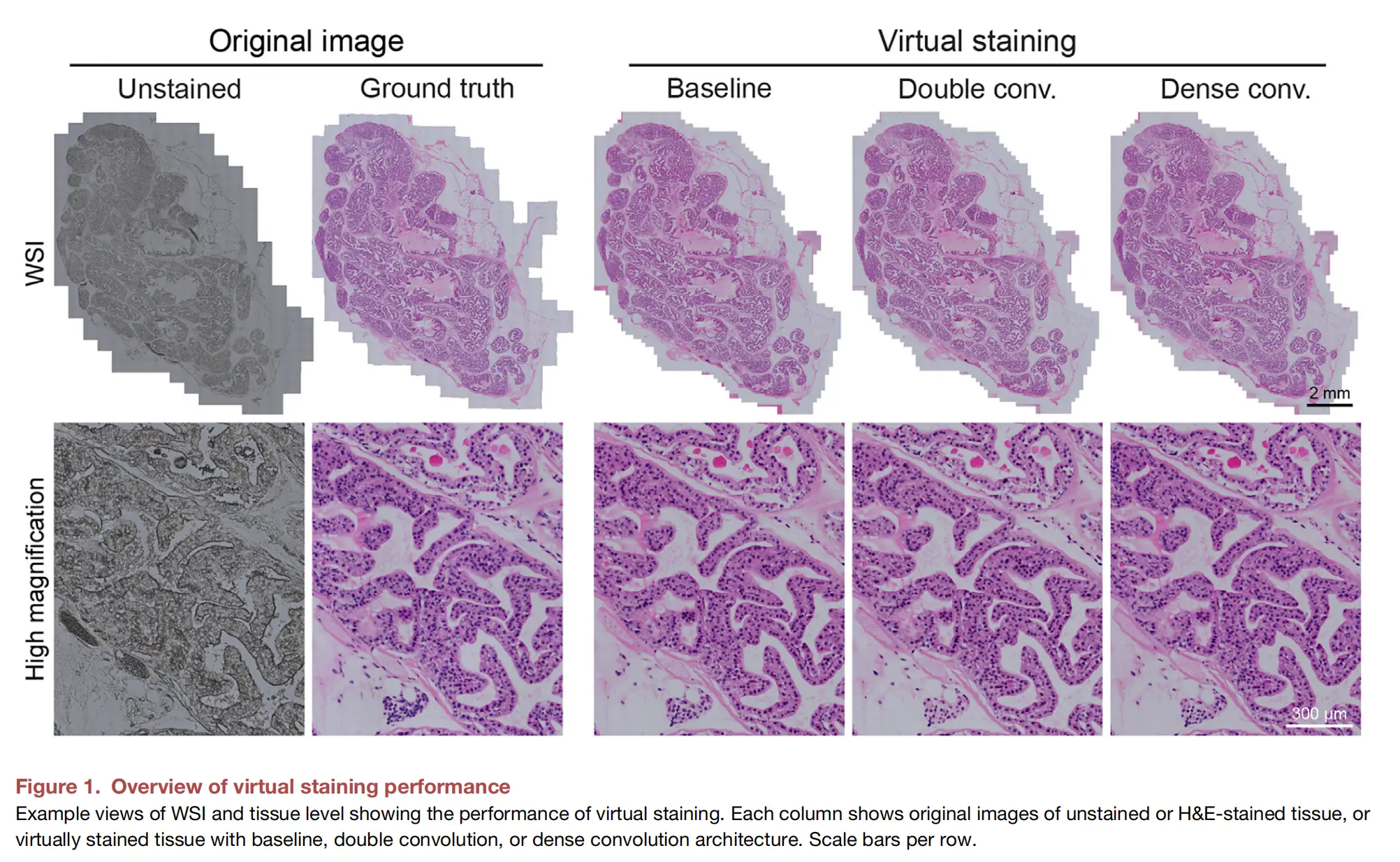
Figure 1提供了对虚拟染色性能的概览,展示了整个切片图像(WSI)和组织层面的示例视图,比较了原始未染色或H&E染色的组织图像与使用基线(baseline)、双重卷积(double convolution)和密集卷积(dense convolution)架构的虚拟染色图像的性能。

以下是对Figure 1的分析:
图像布局:该图将每一列都展示为不同的染色状态,包括原始未染色、H&E染色以及三种不同神经网络架构生成的虚拟染色图像。
染色状态比较:每一列中的图像可以直观地比较不同染色状态下的组织细节。未染色的组织图像几乎没有颜色,而H&E染色图像则展示了细胞和组织结构的颜色细节。
虚拟染色架构:
基线(Baseline):使用pix2pix模型的标准实现,可能在某些细节上不如其他两种架构清晰。双重卷积(Double Convolution):在编码器和解码器中增加了卷积层的数量,可能在细节捕捉上有所改进,但可能仍然存在一些伪影或细节丢失。密集卷积(Dense Convolution):采用DenseU-net风格的编码器和解码器,这种架构在增加网络容量方面更为复杂,可能在细节再现和减少伪影方面表现最佳。
尺度条:每行图像旁边都有尺度条,用于指示图像中结构的实际大小,便于比较不同图像间的尺寸比例。
组织细节:通过比较不同架构的虚拟染色图像,可以观察到它们在复制组织细节(如细胞核、细胞质和其他结构)方面的能力。
颜色再现:H&E染色中,细胞核通常呈现为紫蓝色,而细胞质和基质呈现为不同程度的粉红色。虚拟染色图像需要在颜色再现上接近这种效果。
性能评估:通过视觉比较,可以评估不同虚拟染色方法在复制H&E染色特征方面的效果,包括颜色准确性、结构细节的清晰度以及伪影的多少。
Figure 1是评估虚拟染色技术有效性的重要视觉工具,它使研究人员能够直观地看到不同神经网络架构对染色效果的影响。
十、利用对抗学习模型,在病理学图像处理中实现从H&E染色图像到Ki-67染色图像的转换

一作&通讯
| 角色 | 姓名 | 单位(中文翻译) |
|---|---|---|
| 第一作者 | Shuting Liu | 清华大学深圳国际研究生院 |
| 第一作者(共同) | Baochang Zhang | 慕尼黑工业大学计算机辅助医疗程序(CAMP) |
| 通讯作者 | Tian Guan | 清华大学深圳国际研究生院 |
| 通讯作者 | Yonghong He | 清华大学深圳国际研究生院 |
文献概述
这篇文章是关于一种新的对抗学习方法,用于在病理学图像处理中实现从H&E染色图像到Ki-67染色图像的转换。
这种方法特别适用于低资源地区,那里很难进行免疫组化(IHC)检查。
提出了一种名为PC-StainGAN的新型对抗学习方法,该方法利用结构相似性约束和跳跃连接来提高结构细节的保留,并首次提出病理一致性约束和病理表征网络,以确保生成的图像与源图像在不同的染色领域中具有相同的病理属性。
在两个不同的未配对组织病理学数据集上验证了该方法的有效性,并通过大量实验表明,该方法的性能显著优于现有技术。
该方法在未平衡数据集上也表现出稳定和良好的性能,显示出强大的鲁棒性。
论文还讨论了使用深度学习在未配对图像到图像转换领域的潜力,并分析了Cycle-GAN在病理图像分析中的局限性。
论文的实验部分使用了两个数据集来评估所提方法的性能,包括神经内分泌肿瘤数据集和乳腺癌数据集,并通过定量和定性的结果展示了所提方法的高性能和鲁棒性。
论文还探讨了所提方法在临床虚拟染色中的潜在应用,并提供了相关代码的链接。
文章强调了PC-StainGAN在临床应用中的潜力,尤其是在资源受限的环境中,它可以作为一种有效的、节省成本的方法来辅助癌症诊断。
重点关注
Fig. 5 展示了不同方法在图像重建与源图像之间的距离图。

从左到右的列分别对应:
(a) 原始图像:这是用作参考的原始 H&E 染色图像。(b) Cycle-GAN:展示了使用 Cycle-GAN 方法重建的图像与原始图像之间的距离图。颜色越深表示距离越小,即重建图像与原始图像越相似。© Cycle-GAN+skip-connection:展示了在 Cycle-GAN 的基础上添加跳跃连接(skip connection)后的重建图像与原始图像之间的距离图。跳跃连接有助于改善网络对结构信息的传递和重建。(d) Cycle-GAN+SSIM constraint:展示了在 Cycle-GAN 的基础上添加结构相似性(SSIM)约束后的重建图像与原始图像之间的距离图。SSIM 约束有助于网络在重建过程中保持与原始图像的结构相似性。(e) Cycle-GAN+skip-connection+SSIM constraint:展示了同时添加跳跃连接和 SSIM 约束后的重建图像与原始图像之间的距离图。这种组合预期会提供最佳的结构细节保留。
分析这些距离图时,我们可以观察颜色的分布来评估每种方法的性能:
颜色越深(尤其是蓝色),表示重建图像与原始图像的差异越小,结构细节保留得越好。如果某些区域颜色较浅或有不均匀的颜色分布,这可能意味着重建过程中丢失了一些细节,或者存在结构上的偏差。
从实验结果可以预期,(e) Cycle-GAN+skip-connection+SSIM constraint 的距离图将展示出最接近原始图像的结构细节,表明结合跳跃连接和 SSIM 约束的方法在重建图像时能最有效地保留原始图像的结构信息。
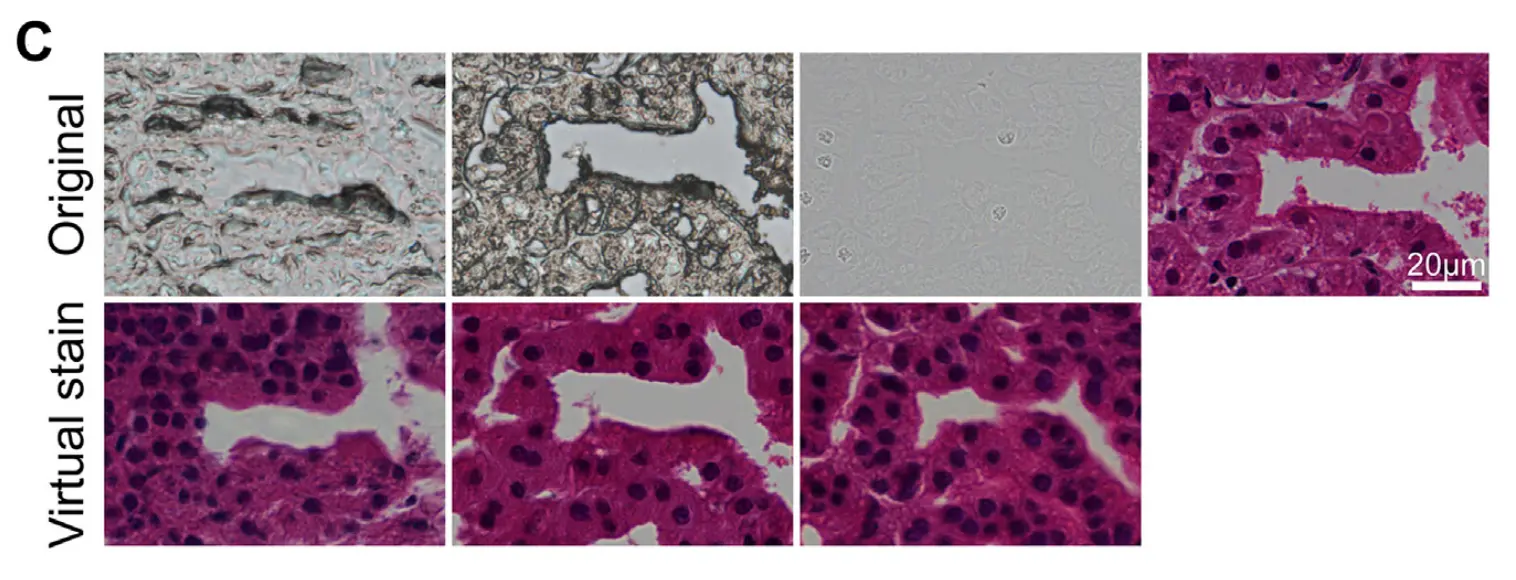
十一、使用未染色组织成像和虚拟HE染色技术进行组织学全切片图像分析

一作&通讯
| 角色 | 姓名 | 单位(中文) |
|---|---|---|
| 第一作者 | Sonja Koivukoski | 东芬兰大学生物医学研究所 |
| 通讯作者 | Leena Latonen | 芬兰癌症研究所 |
文献概述
这篇文章是一篇关于使用未染色组织成像和虚拟HE染色技术进行组织学全切片图像(Whole Slide Images, WSI)分析的研究论文。
研究背景:
组织学是评估组织结构、表型和病理状况的基础方法,通常涉及使用化学染色使透明的组织切片可见。化学染色虽然快速常规,但会永久改变组织并常消耗有害试剂。为了结合测量,需要技术提供基本组织结构的视觉信息,允许从完全相同的组织切片进行额外测量。
研究目的:
开发一种使用未染色组织成像的计算HE染色方法。
研究方法:
使用无监督深度学习(CycleGAN)和前列腺组织切片的全切片图像,比较了在不同条件下成像的组织切片(如石蜡中、空气去石蜡和介质中去石蜡)的性能。测试了3至20微米不同厚度的组织切片。
研究结果:
较薄的切片在提供可虚拟染色复制的信息方面表现更好。石蜡中的组织成像和去石蜡后的组织为虚拟HE染色图像提供了良好的整体组织表示。使用pix2pix模型(一种有条件的对抗网络变体,用于监督图像到图像的转换)显示,通过监督学习和像素级真实值,可以明显改善整体组织形态学的再现。
研究结论:
虚拟HE染色可以用于多种组织,并且可以与20倍和40倍的成像放大倍率一起使用。虽然虚拟染色的性能和方法需要进一步开发,但研究提供了整个未染色切片显微镜检查作为快速、廉价、可行的方法的证据,以产生组织形态学的虚拟染色,同时保留完全相同的组织切片,准备用于后续方法的单细胞分辨率的后续利用。
文章还包括了详细的实验方法、统计评估、模型训练持续时间、组织学评估和讨论等部分。
重点关注
Figure 4 展示了使用不同处理方法的组织切片进行无监督虚拟HE染色的组织学性能,并将其与原始未染色图像和HE染色参考图像进行了比较。
以下是对图表的分析:

(A) WSI级别的低倍镜视图:
这个视图提供了一个宏观的视角,观察整个组织切片(Whole Slide Image, WSI)。低倍镜下可以观察到整个前列腺组织切片的概貌,包括腺体结构的分布和周围基质。该图像用于评估整体组织结构和是否存在明显的病理变化。

(B) 前列腺腺体结构的视图:
这个视图聚焦于前列腺的一个特定腺体结构。可以观察到腺体的形态,包括腺体的大小、形状以及腺体上皮细胞的排列。该图像有助于评估腺体结构的完整性和是否存在异常,如肿瘤或炎症。

© 前列腺上皮细胞的高倍镜视图:
高倍镜下可以详细观察到上皮细胞的形态特征,包括细胞的大小、形状、核的位置以及细胞边界。这个视图对于识别细胞级别的变化非常重要,比如细胞异型性或核的多形性,这些可能是癌症或其他病理状况的迹象。通过与HE染色的参考图像比较,可以评估虚拟染色技术在复制实际染色特征方面的准确性和可靠性。
整体而言,Figure 4 强调了虚拟HE染色技术在复制传统HE染色特征方面的能力,特别是在不同组织学层面上的表现。通过比较原始未染色图像和虚拟染色图像,研究者可以评估虚拟染色技术的性能,并探索其在临床和研究领域的潜在应用。
声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。