药物人必备科研技能!CADD计算机辅助药物设计联合AIDD人工智能药物发现+深度学习蛋白质设计
人工智能前沿技术, 2024-07-16 13:01:02 阅读 52
深度学习在蛋白质设计领域的应用可以加速药物研发和生物治疗等领域的研发进程。通过预测蛋白质的结构和功能,研究人员可以更快地筛选出具有潜在治疗价值的药物候选物,从而缩短药物研发周期和降低研发成本。深度学习在蛋白质设计领域的前沿背景和研究优势主要体现在技术突破、数据量增长和跨学科融合等方面。其研究优势包括提高预测准确性、创新设计能力和加速研发进程等。这些优势使得深度学习在蛋白质设计领域具有广阔的应用前景和重要的科学价值。
CADD技术,即计算机辅助药物设计(Computer Aided Drug Design),是在分子生物学、X射线晶体学的发展和计算机科学的迅速崛起的推动下应运而生的。
CADD技术以量子力学和分子力学的分子模拟理论为基础,通过模拟、计算或模型学习,预测药物与靶标分子之间的作用,从而筛选、设计和优化先导化合物。这种技术在新药研发中具有显著的优势,主要包括且不限于提高药物设计命中率、增加成功率、降低研发成本、缩短研发周期
AIDD,即人工智能驱动的设计与发现(AI-Driven Drug Discovery and Design),是一种结合了人工智能(AI)和药物发现与设计的创新技术。近年来,随着计算能力、机器学习和人工智能的进步,AIDD已经变得越来越复杂,并在新药研发中发挥着越来越重要的作用。AIDD技术的优势体现在以下方面:提高效率、降低成本、预测准确性、安全性评估。AIDD技术为药物发现和研发带来了革命性的变革,其独特的优势正在逐渐改变着传统的药物研发模式。
药物两大前沿课程目录
一、深度学习蛋白质设计
二、CADD计算机辅助药物设计
三、AIDD人工智能辅助药物发现
一、深度学习蛋白质设计
课程目标
基于深度学习的通用型蛋白设计模型近几年来发展迅速,从蛋白结构的预测与优化到蛋白的从头设计进行深度教学,本课程从零基础开始讲解,对基础知识进行详细讲解,并且会结合前沿文献讲解相关技术的应用。了解蛋白质设计的底层逻辑与基本规则,并掌握蛋白质设计中的常见蛋白质设计算法的实际操作,具备基本的蛋白质设计算法开发的基础能力及前沿视野。
结构预测基础: 学生将学会通过生物信息学工具分析蛋白质序列,预测其二级结构和三维结构,并理解结构与功能之间的关联。
模型应用与评估: 学生将能够使用机器学习和深度学习模型进行蛋白质结构预测,同时学习如何评估模型的准确性和可靠性,选择正确的工具加以应用。
药物设计: 掌握与蛋白质结构预测相关的药物设计原理,学会设计靶向特定蛋白质的药物分子与多肽药物分子,理解蛋白类药靶相互作用的机制。
授课老师
主讲老师们来自国内最顶尖高校,组内工作主要聚焦新型蛋白质设计与蛋白质结构预测算法开发与自动化药物高通量筛选方向,已在Nature Chemistry子刊, JCAS,Angew,JMC,JCTC等权威期刊上发表SCI检索论文20余篇
课程内容
向下划动查看全部内容
第一天 Python编程基础
1. Python 基础
1.1Python 简介:了解Python的历史、特点和与其他编程语言的比较。
1.2安装和设置环境:安装Python,设置Python开发环境(如Anaconda、Jupyter notebook)。
1.3基本语法:数据类型(整数、浮点数、字符串、布尔值)、变量、基本运算符。
1.4控制结构:条件语句(if-else)、循环语句(for循环、while循环)。
1.5函数:定义函数、参数、返回值、作用域、递归。
1.6数据结构:列表、元组、字典、集合、操作和常用方法。
1.7文件操作:读写文件,文件与异常处理。
2. Python 进阶
2.1类和对象:面向对象编程基础,创建类,实例化对象,理解封装、继承和多态。
2.2模块和包:导入标准模块,使用第三方包,创建自定义模块和包。
2.3高级功能:列表推导式、生成器、迭代器、装饰器和匿名函数。
3. Python 在科学计算中的应用
3.1NumPy:数组创建、数组操作、数学计算、线性代数等。
3.2Matplotlib:基础图表、科学图形、图表定制。
4. 数据分析与可视化
4.1Pandas:数据结构(Series、DataFrame)、数据载入、数据清洗、数据统计、数据合并。
4.2数据可视化:使用 Matplotlib 和 Seaborn 进行高级数据可视化。
5. 蛋白质设计中的特定应用
5.1BioPython:序列处理、数据库访问、生物学数据的分析。
5.2脚本编写:自动化常见的蛋白质设计任务,如序列对比、结构预测。
5.3机器学习:使用Scikit-learn进行特征提取、模型训练、评估和优化。
6. 实战项目
61项目1:蛋白质序列数据分析,如统计特定序列的频率、可视化序列分布等。
6.2项目2:蛋白质结构预测,使用机器学习技术预测蛋白质的二级结构或功能位点。
6.3项目3:开发一个小型的蛋白质设计工具,集成数据处理、分析及可视化功能。
第二天 shell命令行操作基础
1. Shell环境简介
1.1什么是Shell:了解Shell是什么,以及它如何与操作系统交互。
1.2不同类型的Shell:Bash, Zsh, Tcsh的介绍。
1.3访问Shell:如何打开终端窗口,基础的命令行界面操作。
2. 基础命令
2.1文件系统操作:cd, ls, pwd, mkdir, rm, touch 等命令的使用。
2.2文件操作:cat, more, less, head, tail, grep, find等命令。
2.3权限和所有权:使用chmod, chown, chgrp改变文件的权限和所有权。
2.4文本处理:echo, cat, cut, sort, uniq, tr, awk, sed等工具的基本使用。
2.5归档和压缩:tar, gzip, gunzip, zip, unzip等命令。
3. Shell脚本编写
3.1创建和执行Shell脚本:如何编写一个简单的脚本并使其可执行。
3.2变量和数据类型:学习如何在脚本中定义和使用变量。
3.3流程控制:
3.4条件语句:if, else, elif, case等语句的使用。
3.5循环结构:for, while, until循环的使用。
3.6函数:如何定义和使用函数。
3.7输入和输出:处理用户输入和脚本输出。
3.8引用和转义字符:学习如何在命令行中正确使用单引号、双引号和转义字符。
4. 高级Shell编程
4.1调试Shell脚本:如何调试Shell脚本,包括设置和使用调试选项。
4.2正则表达式:基本正则表达式的应用,结合grep, sed, awk使用。
4.3环境管理:了解PATH和其他环境变量的作用和管理方法。
4.4脚本的安全性:编写安全的脚本以避免常见的安全问题。
5. 实用案例和项目
5.1数据备份脚本:创建一个自动备份你的重要文件的脚本。
5.2文件整理脚本:编写一个自动整理下载文件夹中文件的脚本。
5.3PDB文件分析脚本的编写
第三天 蛋白质设计基础: 从经典力场到深度学习
1. 如何计算蛋白质构象的能量?
a) 蛋白质可视化与编辑常用方法
i. pymol使用方法简介
ii. chimera使用方法简介
iii. pdb文件格式详解
iv. 使用python biopython、pymol等库编辑蛋白质结构
b) 分子力学、溶剂化能简介
i. 分子力学公式形式
ii. 溶剂化能的计算方法
iii. MM/PBSA方法计算结合自由能
2. 基于统计势函数的蛋白质设计方法——Rosetta
a) 统计势函数的一般定义
b) 蛋白质设计中的统计势函数
i. Rosetta统计势定义
ii. Rosetta能量函数常见项及物理意义
c) 基于Rosetta势函数的蛋白设计
i. 设计流程
ii. 实验结果
3. 蛋白-蛋白对接的强大——无先验知识的蛋白质药物设计流程
a) 蛋白-蛋白对接简介
i. 蛋白-蛋白对接的定义
ii. RifGen对接方法介绍
b) 蛋白质药物设计
i. 设计流程
ii. 实验结果
4. 深度学习强势登场——蛋白质设计模型ProteinMPNN
a) MPNN 消息传递神经网络简介
b) ProteinMPNN模型简介
i. 模型结构介绍(输入、输出、参数……)
ii. 模型使用(主编程语言Python)
c) 基于ProteinMPNN的蛋白质设计
i. 设计流程
ii. 实验结果
5. 平分秋色还是天壤之别?两种蛋白设计方法的比较
a) 深度学习模型具有更高的序列恢复率
b) 深度学习模型可实现rosetta、alphafold不可完成的设计任务
c) 深度学习模型的短板
第四天 Alphafold vs Rosettafold
1.蛋白结构预测背景介绍
2.早期蛋白质结构预测算法:从统计分析到深度残差网络
2.1 直接耦合分析和互信息计算
2.2 深度残差网络和蛋白质接触图预测
2.3 蛋白质距离矩阵预测
3. 几何约束的梯度下降法到端到端深度学习的蛋白结构预测
3.1 trrosetta和alphafold简介
3.2 端到端几何深度学习方法介绍
4. AF和AF2的差异与创新
4.1 第一代Alphafold简介
4.2 Alphafold2详解
5. Rosettafold详解
5.1 SE3网络
第五天 基于Alphafold的下游应用
1. AF2多体蛋白结构预测的关键问题与解决途径
1.1 多序列比对中序列拼接配对问题
1.2 模板匹配问题
2. 利用AF2做蛋白和多肽柔性对接
2.1蛋白质表面几何物理化学互补问题
2.3 多肽柔性/构象处理
3. 利用AF2做蛋白结构和序列新设计
3.1 trrosetta幻想设计
3.2 AF2序列和结构幻想设计
4. 利用AF2做结构聚类发现新结构和功能
4.1 alphadatabase数据库结构简介与分析
4.2 foldseek结构比对工具介绍
4.3 新结构与新功能
5. 利用AF2做多构象预测和功能发现
5.1 MSA采样聚类分析及结构预测
5.2 不同MSA可以预测构象之间的转变和功能
6. 利用AF2的部分算法模块做模型质量评估和侧链构象等
6.1 三角机制提升蛋白质模型质量评估
6.2 局部三角机制和evoformer用于蛋白质侧链预测
第六天 蛋白质的从头生成模型
1. Rosettafold的基本架构回顾
2. 基于Rosettafold的改进
a) 有关扩散模型
b) 基于扩散模型的模型修改
3. RFdiffusion实现通用性蛋白结构生成
a) 蛋白质binder生成
b) 基于骨架结构的蛋白质结构生成
c) 蛋白质单体的从头生成
d) 多聚体蛋白的从头生成
4. ProteinGenerator实现蛋白质骨架与序列的co-design
a) 隐空间中的蛋白质序列-结构的联合分布
b) 与rfdiffusion的异同
5. Rosettafold AA实现多类生物大分子结构预测与生成
a) 加入小分子结构预测器
b) 将局部坐标系迁移到小分子结构
6. Chroma的基本构架与实现
a) 模型讲解
b) 利用chroma逼近蛋白构象空间全空间采样与生成
第七天 大语言模型在蛋白质设计中的应用及多肽设计
1. ProGEN的基本实现
a) 模型构架讲解
b) 与基于结构方法的比较
2. ProGEN的性能与改进
3. ESM-fold的基本构架
a) ESM网络构架
b) ESMfold网络讲解
c) 与alphafold方法的对比
4. ESM-fold的性能评估
5. 基于大语言模型的蛋白质生成模型的下游应用
a) 蛋白质结构的快速预测
b) 大型蛋白复合物结构预测
6. 多肽设计与蛋白质设计的区别与联系
a) 分子结构稳定性
i. 二级结构含量
ii. 氢键网络与局部二级结构
b) 分子的功能性异同
c) 蛋白质设计方法在多肽设计面临的瓶颈
7. 多肽设计算法
a) 基于Rosetta实现多肽设计
i. Flexpepdock
ii. Anchor extension
b) 基于RF diffusion实现多肽设计
i. 参数的设定与优化方案
ii. 生成结构的评估
c) 基于alphafold 梯度下降进行多肽骨架和序列设计
d) 多肽对接算法
i. 基于Autodock 的多肽对接
ii. 基于alphafold的多肽柔性对接
iii. 其余对接算法
8. 基于多肽蛋白复合物训练的深度学习多肽设计算法
二、CADD计算机辅助药物设计
STUDY
课程目标
1.将深入了解计算机辅助药物设计(CADD)的整个研究过程,包括从靶点识别、分子建模到虚拟筛选和后续的实验验证等关键步骤。课程将重点讲授各个阶段中所需的关键技术,并确保能够熟练使用一系列常用的CADD工具。此外,将对这些工具背后的科学原理及其实际操作技巧获得全面的理解。
2.本课程将引导学生探索计算机辅助药物设计与人工智能药物设计(AIDD)之间的联系,并深入讨论两者如何相互促进和补充。学生将了解到近年来CADD领域的最新进展,特别是人工智能技术如何在药物设计领域中发挥作用,以及这些进展对未来研究和应用的潜在影响。
3.随着生物信息学和计算生物学的迅速发展,新兴技术如AlphaFold正在改变传统的药物设计方法。在此课程中,学生将详细了解这些技术的工作原理和应用领域,并对其潜在局限性有所认识。课程将通过实例讲解如何有效地利用这些技术进行药物研发。
4.为了让学生能够紧跟行业发展的步伐,本课程将介绍CADD领域中的最新应用案例和研究成果。学生将通过分析最新的研究论文和案例研究,理解当前CADD技术如何在实际药物研发中发挥作用,以及这些进展如何推动药物设计领域向前发展。
授课老师
CADD主讲老师主讲吴老师来自国内顶尖985高校,组内工作主要聚焦新型药物设计、分子对接算法开发与自动化药物高通量筛选方向,已在PNAS,JMC,JCTC,JCIM等权威期刊上发表SCI检索论文15余篇
向下划动查看全部内容
第一天、药物设计基础
上午
背景与理论知识以及工具准备
1.课程背景简介
1.1药物设计简介
1.2基于结构靶向药物设计(SBDD)简介
1.3CADD简介
1.4CADD研究流程
1.5CADD与AIDD
2.靶点与配体构建
2.1靶点信息获取
2.1.1 PDB数据库
2.1.2 Uniprot数据库与AlphaFold DB
2.1.3 ESMfold与ESM Altas
2.1.4 AlphaFold 3原理
2.1.5 AlphaFold 3与AlphaFold2区别
2.2配体信息获取
2.2.1常见小分子数据库(Drug Bank、ZINC、ChEMBL等)
2.2.2利用Chemdraw手绘分子
下午
一般的蛋白-配体分子对接讲解
1.对接的相关理论介绍
1.1分子对接的概念及基本原理
1.2分子对接的基本方法
1.3分子对接的常用软件
1.4分子对接的一般流程
2.常规的蛋白-配体对接
2.1收集受体与配体分子
2.2复合体预构象的处理
2.3准备受体、配体分子
2.4蛋白-配体对接
2.5对接结果的分析
2.6 对接结果可视化(以pymol为例)
2.6.1 pymol基本操作
2.6.2 相互作用绘制
2.6.3 pymol出图的设置
2.6.4 pymol wiki与pymol进阶
以新冠病毒蛋白主蛋白酶靶点及相关抑制剂为例
第二天
虚拟筛选
1.linux操作系统简介
1.1linux系统基本命令介绍
1.2bash脚本批量化处理介绍
2.药物ADMET预测
3.基于GPU加速的分子对接工具
4.结果分析
第三天
CADD工具进阶
1.柔性对接
1.1基于物理模型的柔性对接
1.2基于AI的柔性对接
2.蛋白-蛋白对接
2.1基于物理模型的蛋白-蛋白对接
3.金属配体对接
4.共价对接
5.核酸对接
5.1RNA结构预测工具
5.2RNA对接工具
6.RF-AA与AlphaFold 3
第四天
分子动力学模拟(linux与gromacs使用安装)
1. linux系统的介绍和简单使用
1. 分子动力学的理论介绍
2. 分子动力学相关程序
2.1 CHARMM
2.2 Amber
2.3 Gromacs
2.4 openMM
2.5 NAMD
3. 相关力场介绍
4. MDtraj包的介绍与使用
第五天
蛋白-配体分子动力学模拟的执行
1.蛋白-配体在分子动力学模拟的处理流程
2.蛋白晶体的准备
3.蛋白-配体模拟初始构象的准备
4.配体分子力场拓扑文件的准备
4.1 高斯的简要介绍
4.2 ambertools的简要介绍
4.3生成小分子的力场参数文件
5.对复合物体系温度和压力分别限制的预平衡
6.无限制的分子动力学模拟
7.结合自由能mm-pbsa/mm-gbsa计算
8.轨迹后处理及分析
以新冠病毒蛋白主蛋白酶靶点及相关抑制剂为例
第六天
分子动力学进阶
1.膜蛋白分子动力学模拟
1.1膜蛋白数据库
1.2蛋白跨膜区域预测
1.3CHARMM-GUI使用
2.增强采样技术
2.1REMD与REST/REST2
2.2Plumed安装与使用
2.3aMD与GaMD
2.4基于AI的增强采样技术
第七天
变构蛋白与蛋白系综
1.变构药物
1.1变构蛋白简介
1.2变构蛋白数据库
1.3变构位点预测
1.4变构通路计算
2.蛋白系综
2.1蛋白系综分析
2.2蛋白系综预测
2.3靶向天然无规蛋白的药物设计
部分模型案例图片:
三、AIDD人工智能辅助药物发现与设计
课程目标
目标:了解人工智能辅助药物发现和设计的前沿,学习人工智能模型,熟悉工具包的安装与使用,掌握一定的算法编程能力,能够运用计算机方法研究药物相关问题。通过大量的案例讲解和实践操作,具备一定的AIDD模型构建和数据分析能力。学习线路:1. 基础数学知识:学习数理统计、概率论和微积分等基础知识。这些数学工具在AIDD中起到至关重要的作用,用于处理和分析大量数据,以及构建和优化算法模型。2. 机器学习基础:掌握机器学习的基础理论和方法,包括决策树、随机向量机等传统机器学习模型,以及深度神经网络、卷积神经网络、循环神经网络等深度学习算法。理解这些模型的原理和应用场景,为后续的药物发现与设计提供技术支持。3. 药物化学与生物学知识:学习药物化学和生物学的基础知识,了解药物的结构、性质以及与生物体的相互作用。4. AIDD专业课程:深入学习AIDD的专业课程,包括理化性质预测、代谢预测、转运体预测以及化合物设计等内容。通过实践案例和项目,掌握AIDD的实际应用方法和技巧。5. 编程技能:学习Python等编程语言,掌握数据结构与算法,以及相关的机器学习库和框架。这些编程技能对于实现AIDD算法、处理和分析数据是必不可少的。6. 实践经验:参与实际的药物发现与设计项目,积累实践经验。通过与其他领域的专家合作,了解AIDD在实际应用中的挑战和解决方案。
了解人工智能辅助药物发现和设计的前沿,学习人工智能模型,熟悉工具包的安装与使用,掌握一定的算法编程能力,能够运用计算机方法研究药物相关问题。通过大量的案例讲解和实践操作,具备一定的AIDD模型构建和数据分析能力。
授课老师
AIDD主讲老师李老师,副教授,省级拔尖青年人才,硕士生导师,专业负责人。长期从事以蛋白质为主要研究对象的人工智能和生物医药交叉研究。主持科研基金国家级3项、省部级4项。以第一或通讯作者身份在J. Chem. Inf. Model、Comput. Biol. Med、BMC Bioinform等计算生物学权威期刊上发表学术论文20余篇。
向下划动查看全部内容
第一天、AIDD及编程基础
1. AIDD简介
(1) 药物发现与设计
(2) 基于配体的方法
(3) 基于结构的方法
2. 综合数据库
(1) 蛋白质
① 结构数据库PDB
② 结构显示Pymol
③ 序列数据库uniport
(2) 药物/小分子
① PubChem、ChEMBL
② Python批量化处理RdKit
(3) 相互作用
① PDBbind
② 结合位点的分析
3. 编程环境
(1) 管理第三方包:anaconda
(2) 集成性编程环境:pycharm
(3) 交互式编程环境:ipython notebook
4. 第三方库基本使用方法
(1) Numpy
① 数组、切片
② 简单的数据处理
(2) Pandas
① 表格化
② 矩阵操作
③ 大型数据分析
(3) Matplotlib
① 绘图
② 集成环境seaborn
(4) Pytorch:深度学习框架
5. 数据降维及方法
(1) 主成分分析(PCA)
(2) 独立分量分析(ICA)
(3) 等距特征映射(ISOMAP)
(4) T 分布随机近邻嵌入(t-SNE)
第二天人工智能基础
1. 机器学习的一般流程
(1) 定义函数
① 函数形式:输入变量、线性、非线性
② 具有可学习参数的函数
(2) 转换求解——损失函数:定义、实现、变体
(3) 迭代优化——梯度下降算法
2. 经典模型
(1) 线性及非线性映射:线性回归、逻辑回归
(2) 聚类:K近邻聚类
(3) 核方法:支持向量机
(4) 树方法:决策树、随机森林
(5) 神经网络:多层感知机
(6) 实验:scikit-learn基本调用
3. 卷积神经网络
(1) 卷积:卷积操作的基本流程、感受野、卷积可解释性
(2) 池化:维度变换的意义
(3) 3D卷积神经网络
4. 语义模型
(1) 循环神经网络:长程短记忆单元
(2) 序列对序列:端对端的序列映射
(3) 注意力机制:查询、键、值
(4) Transformer
5. 生成式模型
(1) 自编码机:编码和解码
(2) 对抗生成网络:生成器和辨别器
(3) 自监督学习
6. 对比学习
(1) 数据增强
(2) 对比损失
6.领域论文精讲
第三天、图神经网络
1. 图建模
(1) 图的基本概念
(2) 节点和边的特征表示
(3) 图、节点、边任务
2. 消息传播机制
(1) 邻居聚合:聚合函数、更新函数
(2) 常见聚合函数:GCN、GraphSAGE、GIN、GAT
(3) 图池化:最大、最小、平均、求和、层次化
3. 图神经网络集成框架
(1) PyG
(2) DGL
(3) 实验:PyG基本用法
4. 几何向量感知机
(1) 标量和向量
(2) 空间旋转不变性
(3) 几何向量融合
5. 图对比学习
(1) 图增强
(2) 对比损失
5.论文精讲
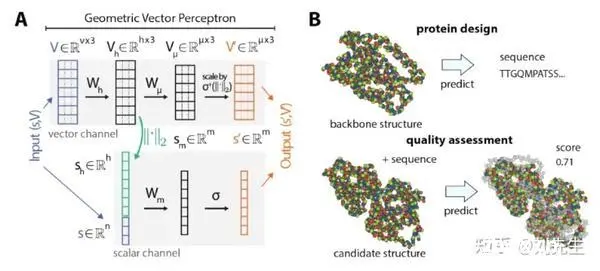
第四天、药物靶标相互作用建模
1. 药物靶标的基础知识
(1) 靶标的定义和分类
(2) 药物与靶标的结合方式
(3) 靶标的生理作用
2. 药物靶标相互作用理论模型
(1) 直接相互作用
① 锁-钥模型
② 配体诱导模型
③ 构象选择模型
(2) 间接相互作用
① 信号转导通路
② 蛋白质-蛋白质相互作用
3. 药物靶标的数据表示和特征提取
(1) 小分子的一般表示
① SIMLES、二维分子、三维结构
② 分子描述符:分子指纹
③ 基于分子特征的聚类
(2) 蛋白质的一般表示
① 序列、结构
② 结构图
(3) 特征选择与降维
4. 药物靶标亲和性预测
(1) 序列与序列建模
(2) 序列和结构建模
(3) 结构和结构建模
5. 模型解释与可解释性
(1) 可解释性模型的应用
(2) 解释性特征分析
6.案例论文精讲(两篇)
第五天、基于空袋的药物设计
1. 蛋白质结构与活性口袋
(1) 蛋白质的结构层级
(2) 口袋在药物设计中的作用和重要性
(3) 活性口袋的定义和特征
(4) 不同类型活性口袋的示例
2. 口袋的结构与功能
(1) 口袋的构成元素
(2) 口袋与配体的相互作用
(3) 口袋结构的动态性和可塑性
3. 口袋信息的表示与分析
(1) 口袋的定位
(2) 表面三角剖分
(3) 几何描述符
(4) 化学描述符
4. 蛋白质结构微环境
(1) 微环境的概念和分类
(2) 微环境的几何表征
(3) 微环境的物理化学特性分析
5. 基于口袋微环境的药物设计
(1) 药物与口袋微环境的相互作用
(2) 药物与口袋微环境的结合模式
(3) 药物设计中的口袋微环境优化策略
(4) 结合口袋微环境分析进行药物筛选与优化
(5) 案例分析
6.案例论文精讲(两篇)
特色
线上授课时间和地点自由,建立专业课程群进行实时答疑解惑,理论+实操授课方式结合大量实战案例与项目演练,聚焦人工智能技术在药物设计、蛋白设计领域的最新研究进展,课前发送全部学习资料,课后全程答疑达到即学即用效果;
完全贴合学员需求的课程体系设计,定期更新的前沿案例,由浅入深式讲解,课后提供无限次回放视频,免费赠送二次学习,发送全部案例资料,永不解散的课程群,可以与相同领域内的老师同学互动交流问题,让求知的路上不再孤单!
时间
深度学习蛋白质设计时间:
2024.7.13----2024.7.14全天授课(上午9:00-11:30下午13:30-17:00)
2024.7.17----2024.7.18晚上授课(晚上19:00-22:00)
2024.7.20----2024.7.21全天授课(上午9:00-11:30下午13:30-17:00)
2024.7.27----2024.7.28全天授课(上午9:00-11:30下午13.30-17:00)
腾讯会议 线上授课(共七天授课时间 提供全程回放视频)
CADD计算机辅助药物设计时间:
2024.6.29----2024.6.30全天授课(上午9:00-11:30下午13:30-17:00)
2024.7.01----2024.7.05晚上授课(晚上19:00-22:00)
2024.7.06----2024.7.07全天授课(上午9:00-11:30下午13:30-17:00)
2024.7.09----2024.7.10晚上授课(晚上19:00-22:00)
腾讯会议 线上授课(共七天授课时间 提供全程回放视频)
AIDD人工智能辅助药物发现与设计时间:
2024.7.13----2024.7.14全天授课(上午9:00-11:30下午13:30-17:00)
2024.7.17----2024.7.18晚上授课(晚上19:00-22:00)
2024.7.20----2024.7.21全天授课(上午9:00-11:30下午13:30-17:00)
腾讯会议 线上(共五天时间 提供全程回放视频)
声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。