医学图像分割的基准:TransUnet(用于医学图像分割的Transformer编码器)器官分割
听风吹等浪起 2024-09-20 16:01:01 阅读 81
1、 TransUnet 介绍
TransUnet是一种用于医学图像分割的深度学习模型。它是基于Transformer模型的图像分割方法,由AI研究公司Hugging Face在2021年提出。
医学图像分割是一项重要的任务,旨在将医学图像中的不同结构和区域分离出来,以便医生可以更好地诊断和治疗疾病。传统的医学图像分割方法通常使用基于卷积神经网络(CNN)的方法,但这些方法在处理大量图像数据时可能存在一些限制,如需要大量标注数据、对图像中小目标的分割效果不佳等。
TransUnet通过引入Transformer模型来提高医学图像分割的性能。Transformer模型在自然语言处理中已经取得了显著的成功,它的一个重要特点是能够处理长距离的依赖关系。TransUnet将这种特点应用于图像分割中,通过将图像划分为一系列的图像块,并使用Transformer模型对这些块进行编码和解码,实现对图像中不同结构和区域的准确分割。
TransUnet的核心组件是自注意力机制,它能够学习图像中不同部分之间的关系,并在编码和解码过程中进行信息传递。通过多层自注意力机制的堆叠,TransUnet能够捕捉到图像中的全局上下文信息,并准确地分割出图像中的不同结构和区域。
与传统的基于卷积神经网络的方法相比,TransUnet在医学图像分割任务上取得了显著的性能提升。它能够更好地处理图像中的小目标和复杂结构,减少了对大量标注数据的依赖,并且在不同的医学图像数据集上都取得了优异的效果。
总的来说,TransUnet是一种基于Transformer模型的医学图像分割方法,通过引入自注意力机制和全局上下文信息的传递,提高了医学图像分割的准确性和效率。它为医生提供了一种精确分割医学图像的工具,有助于更好地诊断和治疗疾病
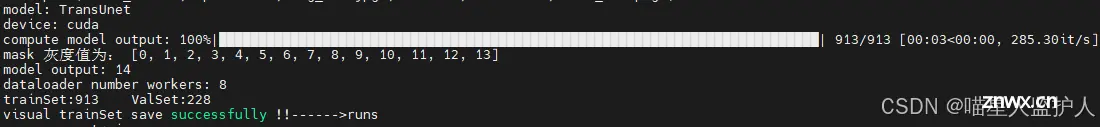
2、 实验1(腹部13器官分割)
TransUnet 多分割项目实验
2.1 数据集介绍
实验使用的数据集是腹部的13器官分类、加上背景0就是14类别的多分割任务
标签如下:
<code>{
"0": "background",
"1": "spleen",
"2": "right kidney",
"3": "left kidney",
"4": "gallbladder",
"5": "esophagus",
"6": "liver",
"7": "stomach",
"8": "aorta",
"9": "IVC",
"10": "veins",
"11": "pancreas",
"12": "rad",
"13": "lad"
}
中文标签:1. 脾脏 2. 右肾 3. 左肾 4. 胆囊 5. 食道 6. 肝脏 7. 胃 8. 主动脉 9. 下腔静脉 10. 门静脉和脾静脉 11. 胰腺 12 右肾上腺 13 左肾上腺

值得注意的是,因为原始的数据是CT数据,对比度较小,这里对数据进行了对比度拉伸,并且全部缩放到512*512的大小。数据的标签是0、1、2、3 ... 12、13的阈值图像
数据集:

标签:
2.2 训练
这里将数据集按照 8:2 比例分成了训练集和验证集

优化器为 optimizer = torch.optim.AdamW(model.parameters(), lr=0.01, weight_decay=0.01)
学习率的衰减策略是余弦退火的cos衰减,batch_size = 8,epoch 设定为100

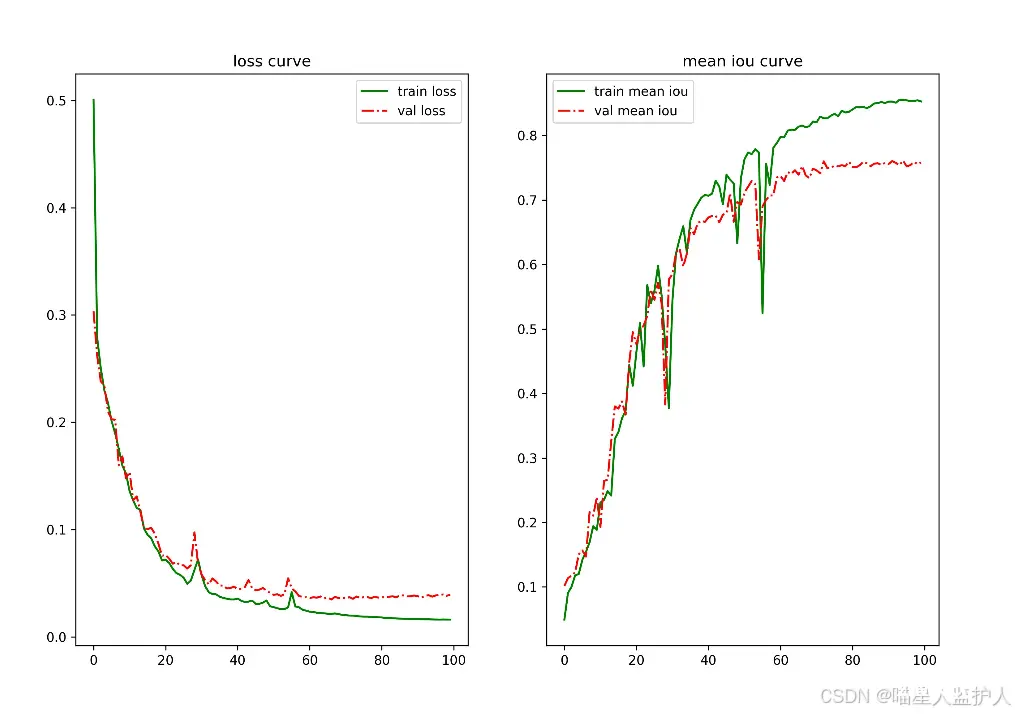
从左到右依次为epoch、train loss、train iou、val loss、val iou

2.3 评估模型
评估模型应该用测试集,因为一开始没有划分测试集,这里用验证集是一样的
这里采用 best.pt
各项指标分别为,这里的列表分别是每个前景的iou、recall等等
global correct: 0.9881
precision: ['0.9930', '0.9653', '0.9447', '0.9456', '0.8626', '0.8249', '0.9715', '0.9547', '0.9285', '0.8862', '0.7844', '0.8250', '0.6903', '0.6778']
recall: ['0.9946', '0.9629', '0.9289', '0.9399', '0.8278', '0.6833', '0.9729', '0.9265', '0.9263', '0.8522', '0.7127', '0.7678', '0.5549', '0.4758']
IoU: ['0.9877', '0.9306', '0.8810', '0.8917', '0.7314', '0.5968', '0.9459', '0.8875', '0.8646', '0.7681', '0.5960', '0.6603', '0.4443', '0.3880']
mean IoU: 0.7553
推理如下,输入数据为:

推理gt:

掩膜效果:
2.4 项目下载
代码、数据集、训练好的权重下载参考:
基于Transformer-Unet实现的对腹部多脏器13类别的分割实战【包含代码+数据集+训练结果】资源-CSDN文库
3、实验2(Synapse多器官分割数据集)
TransUnet 多分割项目实验
3.1 数据集介绍
实验使用的数据集是Synapse multi-organ segmentation dataset(Synapse多器官分割集)
腹部CT扫描 8个腹部器官(主动脉、胆囊、脾、左肾、右肾、肝、胰腺、脾、胃)
标签灰度值从1-7

值得注意的是,因为原始的数据是npz数据,这里输入应该是图像,所以对数据进行的提取,并且全部缩放到512*512的大小。数据的标签是0、1、2、3 ... 7、8的阈值图像


3.2 训练
这里同样将数据集按照 8:2 比例分成了训练集和验证集

优化器为 optimizer = torch.optim.AdamW(model.parameters(), lr=0.01, weight_decay=0.01)
学习率的衰减策略是余弦退火的cos衰减,batch_size = 8,epoch 设定为100

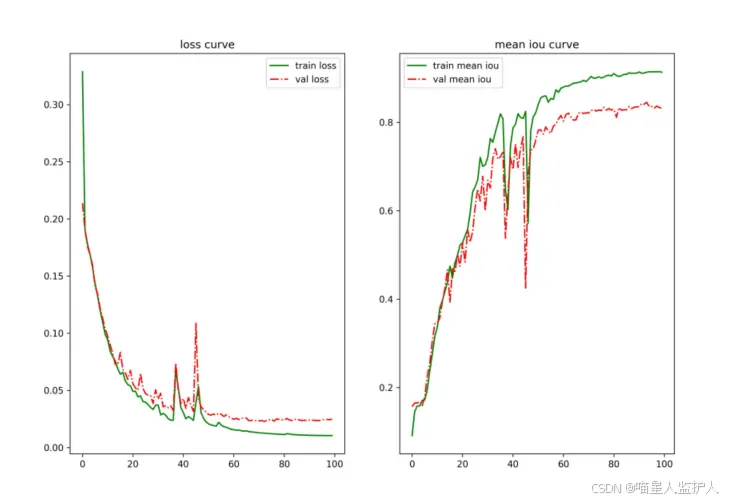
loss和iou曲线为:

从左到右依次为epoch、train loss、train iou、val loss、val iou
3.3 评估模型
指标如下:
global correct: 0.9924
precision: ['0.9958', '0.9089', '0.8571', '0.9012', '0.9353', '0.9670', '0.8367', '0.9643', '0.9299']
recall: ['0.9967', '0.9085', '0.8419', '0.9289', '0.8797', '0.9642', '0.6350', '0.9687', '0.9267']
IoU: ['0.9925', '0.8327', '0.7383', '0.8431', '0.8292', '0.9335', '0.5650', '0.9351', '0.8662']
mean IoU: 0.8373
推理如下:

推理gt:

掩膜效果:

3.4 项目下载
代码、数据集、训练好的权重下载参考:
基于Transformer-Unet实现的对Synapse多器官(8类)分割实战【包含代码+数据集+训练结果】资源-CSDN文库
声明
本文内容仅代表作者观点,或转载于其他网站,本站不以此文作为商业用途
如有涉及侵权,请联系本站进行删除
转载本站原创文章,请注明来源及作者。